References >> PCR Primer
PCR Primer Design Guidelines
Polymerase Chain Reaction (PCR/ポリメラーゼ・チェーン・リアクション)
ポリメラーゼチェーン反応は20世紀における分子生物学のもっとも大きな発明として広く知られるところです。
PCRは、以下の3つのステップで行われます。 変性、アニーリング、伸長。 まず、遺伝物質を変性させ、二本鎖のDNA分子を一本鎖に変換する。 次に、プライマーは一本鎖の相補的な領域にアニールされる。 第三段階として、DNAポリメラーゼの作用により、プライマーが伸長される。 これらのステップはすべて温度に敏感であり、一般的にはそれぞれ94oC、60oC、70oCが選択される。 反応を成功させるためには、優れたプライマー設計が不可欠である。 以下に述べる重要な設計上の考慮点は、高収率で特異的な増幅を行うための鍵である。 なお、表示されている好ましい値は、当社のすべての製品にデフォルトで組み込まれています。
1. プライマーの長さ。 PCRプライマーの最適な長さは18-22bpであると一般に言われている。 この長さは、十分な特異性を得るには十分な長さで、アニーリング温度でプライマーが鋳型に容易に結合するには十分な短さである。
2.プライマーの融点。 プライマーの融解温度(Tm)とは、DNA二重鎖の半分が解離して一本鎖になる温度であり、二重鎖の安定性を示すものであると定義されている。 一般に52-58 oCの範囲の融解温度を持つプライマーが最良の結果をもたらす。 融解温度が65℃以上のプライマーは、二次アニーリングが起こりやすくなります。 プライマーのTmは、配列のGC含有量から推定することができる。
プライマーのTmの計算式:
融解温度Tm(K)={ΔH/ ΔS + R ln(C)}, または融解温度Tm(oC)={ΔH/ ΔS + R ln(C)}
融解温度Tm(oC)={ΔH/ ΔS + R ln(C)}
プライマーは融解温度Tm(K)が最も高く、最も良い値を示すとされている近接熱力学の理論を用いて計算されます。 – 273.15 ここで
ΔH (kcal/mole) : HはEnthalpy。 エンタルピーとは、物質が持っている熱エネルギーの量である。 ΔHはエンタルピーの変化量である。 上式では、ΔHは最近接塩基対のエンタルピー値をすべて足し合わせて求めている。
ΔS (kcal/mole) : Sは系が示す無秩序の量でエントロピーと呼ばれる。 ΔSはエントロピーの変化量である。 ここでは、各近接塩基対のエントロピー値をすべて足し合わせて求めている。
ΔΔS (salt correction) = ΔΔS (1M NaCl )+ 0.0 ΔΔS (1M NaCl )+ 0.0 ΔS (1M NaCl )+ 0.0 ΔS (1M NaCl )+ 0.0 ΔS (2M NaCl )+ 0 ΔS (2M NaCl ) ΔS (1M NaCl )+ 0 ΔS (2M NaCl ) ΔS (2M Maisono ) ΔS (塩分)補正368 x N x ln()
Where
N is the number of nucleotide pairs in the primer ( primer length -1).
is salt equivalent in mM.
That is the nucleotide pair in the primer ( primer length -1).
計算:
= 一価イオン濃度 +4 x 遊離Mg2+.
3. プライマーのアニーリング温度(Primer Annealing Temperature)。 プライマーの融解温度は、DNA-DNAハイブリッドの安定性の目安であり、アニーリング温度を決定する上で重要である。 高すぎると、プライマーとプレートの結合が不十分となり、PCR産物の収量が低下する。 Taが低すぎると塩基対のミスマッチによる非特異的な産物が生じる可能性がある。 ミスマッチ耐性はPCRの特異性に最も強い影響を与えることが分かっている。
Ta = 0.3 x Tm(primer) + 0.7 Tm (product) – 14.9
where,
Tm(primer) = プライマーの融点
Tm(product) = プロダクトの融点
4.GC Content.(GCコンテンツ)。 プライマーのGC含量(全塩基に対するプライマー中のGとCの数)は40〜60%とする。
5. GCクランプ。 プライマーの3’末端から最後の5塩基以内にGまたはC塩基が存在すること(GCクランプ)により、GとC塩基の結合が強くなり、3’末端での特異的結合を促進することができる。 プライマーの3’末端から5塩基以内にGまたはCが3つ以上含まれることは避けるべきである。
6.プライマー二次構造。 分子間や分子内の相互作用によって生じるプライマー二次構造があると、生成物の収量が低下したり、収量が得られなかったりすることがある。 プライマー鋳型のアニーリングに悪影響を及ぼし、増幅に影響を与える。 プライマーが反応に利用しにくくなる。
i) ヘアピン。 プライマー内の分子内相互作用によって形成され、避けるべきである。 最適には3’末端ヘアピンのΔGが-2 kcal/mol、内部ヘアピンのΔGが-3 kcal/molであれば一般に許容される。
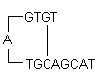
ΔG の定義。 ギブス自由エネルギーGは、一定の圧力で動作するプロセスから抽出することができる仕事の量の尺度である。 反応の自発性の尺度である。 ヘアピンの安定性は、一般に二次構造を壊すのに必要なエネルギーであるΔG値で表される。 ΔGの値が大きいほど、安定で好ましくないヘアピンであることを示す。 3’末端にヘアピンが存在すると、反応に最も悪影響を及ぼす。
ΔΔG = ΔH – TΔS
ii) 自己二量体:プライマーの自己二量体は、プライマーが自身と相同な2つの(同じ意味の)プライマーの間の分子間相互作用によって形成される。 一般にPCRでは、標的遺伝子の量に比して大量のプライマーが使われる。 プライマーが分子間二量体を形成すると、標的DNAとハイブリダイズするよりもはるかに容易に生成物が減少する。 3’末端の自己二量体のΔGは-5 kcal/mol、内部の自己二量体のΔGは-6 kcal/molが最適である。
iii) Cross Dimer: プライマーのクロスダイマーはセンスとアンチセンスのプライマーが相同な場合に分子間相互作用により形成される。 3’末端のクロスダイマーはΔGが-5 kcal/mol、内部のクロスダイマーはΔGが-6 kcal/molであることが望ましい。
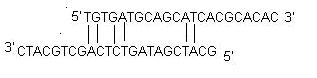
7.反復配列。 リピートとは、何度も連続して出現するジヌクレオチドのことで、ミスプライミングを起こす可能性があるため、避ける必要がある。 例えば ATATATAT。
8.ラン:1塩基のランが長いプライマーはミスプライムする可能性があるため、避けるべきである。 例えば、AGCGGGGGATGGGGは、5と4の塩基’G’のランを持つ。 ランの最大許容量は4bpである。
9. 3′ End Stability。 3’末端から5塩基のΔG値の最大値である。 3’末端が不安定(負のΔGが少ない)であれば、偽プライミングが少なくなる。
10. テンプレートの二次構造を避ける。 一本鎖の核酸配列は非常に不安定で、折りたたまれた構造(二次構造)をとっている。 この二次構造の安定性は、その自由エネルギーと融解温度(Tm)に大きく依存する。 特にqPCRでは、鋳型の二次構造を考慮したプライマー設計が重要である。 アニーリング温度以上でも安定な二次構造上にプライマーを設計した場合、プライマーが鋳型に結合できず、PCR産物の収量に大きな影響を与える。 したがって、PCR反応中に安定な二次構造を形成しない鋳型の領域にプライマーを設計することが重要である。 私たちの製品は、テンプレートの二次構造を見極め、それを避けてプライマーを設計している。 クロスホモロジーを避ける。 プライマーの特異性を高めるためには、相同領域を避けることが必要である。 ある配列に対して設計されたプライマーは、混合物中の他の遺伝子を増幅してはならない。 一般的には、プライマーを設計した後、BLASTを行い、特異性を検証する。 当社の製品は、より良い代替案を提供します。 クロスホモロジーの領域を避けてプライマーを設計することができます。 テンプレートは、適切な非冗長データベースに対してBLASTすることができ、ソフトウェアが結果を解釈します。
プライマーペア設計のパラメータ
1. アンプリコン長。 アンプリコンの長さは、実験の目的によって決まる。 qPCRでは100bp近く、standard PCRでは500bp近くが目標とされる。 鋳型に対する各プライマーの位置が分かっていれば、プロダクトは次のように計算される。 産物の長さ=(アンチセンスプライマーの位置-センスプライマーの位置)+1
2.産物の位置。 プライマーは5’末端、3’末端、または指定された長さ内のどこにでも位置することができる。 一般に、3’末端に近い配列の方が信頼性が高く、最もよく知られている。 溶融温度(Tm)とは、DNA二重鎖の片方が解離して一本鎖になる温度である。 プライマー・テンプレートDNA二重鎖の安定性は、融解温度(Tm)で測定できる。
4.最適アニーリング温度(Ta Opt):プライマー・テンプレートDNA二重鎖は、融解温度(Tm)で測定できる。 Rychlikの公式が最も尊重されている。 当社の製品はこの式で計算し、何千ものお客様がPCRサイクルのアニーリングステップにこの式を使用して良い結果を報告されています。
Ta Opt = 0.3 x(Tm of primer) + 0.7 x(Tm of product) – 14.9
ここで
Tm of primerは安定性の低いプライマーとテンプレートのペアの融点
Tm of productはPCR産物の融点である。
5. プライマーペアのTmミスマッチ計算。 プライマーペアの2つのプライマーはPCR産物の収量を最大にするために融解温度が密接にマッチしている必要がある。
ソフトウェアを用いたプライマー設計
多くのプライマー設計ツールがあり、新規ユーザーも経験者も同様にPCRプライマー設計を支援することができます。
Primer PremierはPCRプライマー設計のために指定されたすべてのガイドラインに準拠しています。 Primer Premier は、単一テンプレート、アラインメント、縮退プライマー設計、制限酵素分析。コンティグ分析、シーケンス用プライマーの設計に使用できます。
qPCR プライマー設計のガイドラインは、若干異なっています。 AlleleID や Beacon Designer などのソフトウェアでは、マルチプレックスアッセイなどの複雑な検出アッセイのためのプライマーやオリゴヌクレオチドプローブの設計、クロスシェアプライマー設計、種特異的プライマー設計、実験コストを削減するためのプライマー設計ができます。
PrimerPlex はマルチプレックス PCR およびマルチプレックス SNP 遺伝子型判定アッセイのためのプライマー設計が可能なソフトウェアです。