B. Replicação do DNA em Bactérias
Em geral, o DNA é replicado pelo desbobinamento da hélice, separação de fios por quebra das ligações de hidrogênio entre os fios complementares, e síntese de dois novos fios por emparelhamento de base complementar. A replicação começa em um local específico no DNA chamado origem da replicação (oriC).
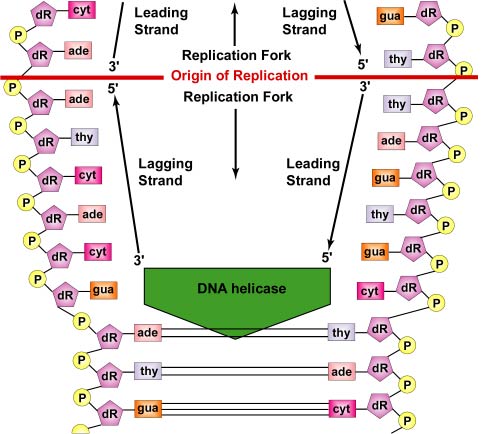
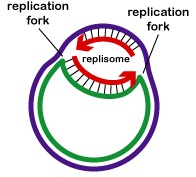
a replicação do ADN é bidireccional a partir da origem da replicação. Para iniciar a replicação de DNA, enzimas desenroladoras chamadas DNA helicases fazem com que segmentos curtos das duas linhas de DNA pai se desenrolem e se separem uma da outra na origem da replicação para formar dois garfos de replicação em forma de “Y”. Estes garfos de replicação são o local real da cópia do ADN (Figura 3). Todas as proteínas envolvidas no agregado de replicação de DNA nos garfos de replicação para formar um complexo de replicação chamado de replisome (Figura \PageIndex{4}}).
Proteínas de ligação de uma só corda ligam-se às regiões de uma só corda para que as duas cordas não se juntem novamente. O desenrolamento da hélice de dupla corda gera super bobinas positivas antes do garfo de replicação. Enzimas chamadas topoisomerases neutralizam isso produzindo quebras no DNA e depois as junta novamente para formar super bobinas negativas a fim de aliviar esta tensão na molécula helicoidal durante a replicação.
As cordas continuam a desenrolar-se e a separar-se em ambas as direcções em torno de toda a molécula de ADN, novas cordas complementares são produzidas pela ligação do hidrogénio dos nucleótidos de ADN livres com os de cada uma das cordas-mãe. À medida que os novos nucleotídeos se alinham em frente a cada cadeia mãe por ligação de hidrogênio, enzimas chamadas polimerases de DNA unem-se aos nucleotídeos por meio de ligações fosfodiéster. Na verdade, os nucleotídeos alinhados por emparelhamento de bases complementares são trifosfatos de desoxinucleotídeos, compostos de uma base nitrogenada, desoxirribose e três fosfatos. Como a ligação fosfodiéster se forma entre o grupo fosfato de 5′ do novo nucleotídeo e o 3′ OH do último nucleotídeo na cadeia de ADN, dois dos fosfatos são removidos fornecendo energia para a ligação (ver Figura 6). No final, cada fita matriz serve como um modelo para sintetizar uma cópia complementar de si mesma, resultando na formação de duas moléculas de DNA idênticas (veja a Figura \PageIndex{7}). Em bactérias, as proteínas Par funcionam para separar cromossomos bacterianos em pólos opostos da célula durante a divisão celular. Elas se ligam à origem da replicação do DNA e puxam ou afastam fisicamente os cromossomos, semelhante ao aparelho mitótico das células eucarióticas. Proteínas Fts, como FtsK no divisoma, também ajudam na separação dos cromossomos bacterianos replicados.
Animação GIF ilustrando a replicação do DNA por par de bases complementares
Na realidade, a replicação do DNA é mais complicada do que isso, devido à natureza das polimerases do DNA. As enzimas DNA polimerase só são capazes de unir o grupo fosfato no carbono de 5′ de um novo nucleotídeo ao grupo hidroxila (OH) do carbono de 3′ de um nucleotídeo já na cadeia. Como resultado, o DNA só pode ser sintetizado na direção de 5′ a 3′ enquanto copia um cordão pai que corre na direção de 3′ a 5′.
Cada cadeia de ADN tem duas pontas. A extremidade 5′ do DNA é aquela com o grupo fosfato terminal no carbono de 5′ da desoxirribose; a extremidade 3′ é aquela com um grupo hidroxil (OH) terminal na desoxirribose do carbono de 3′ da desoxirribose (ver Figura \PageIndex{8}). As duas vertentes são antiparalelas, ou seja, correm em direcções opostas. Portanto, uma das vertentes pai – a que corre de 3′ a 5′ e chamada de vertente principal – pode ser copiada diretamente para baixo em todo o seu comprimento (ver Figura \PageIndex{9}}). No entanto, a outra cadeia pai – a que corre de 5′ a 3′ e chamada cadeia retardada – deve ser copiada descontinuamente em fragmentos curtos (fragmentos de Okazaki) de cerca de 100-1000 nucleotídeos cada um enquanto o DNA se desenrola. Isto ocorre, como mencionado acima, no replisome. O filamento de DNA retardado sai do filamento principal e isso permite que o replissoma se mova ao longo dos dois filamentos, puxando o DNA à medida que a replicação ocorre. É o DNA real, não a DNA polimerase que se move durante a replicação do DNA bacteriano (ver Figura \PageIndex{5}}).
Além disso, as enzimas DNA polimerase não podem iniciar uma nova cadeia de DNA a partir do zero. Elas só podem fixar novos nucleotídeos no grupo 3′ OH de um nucleotídeo em um cordão pré-existente. Portanto, para iniciar a síntese do fio condutor e de cada fragmento de DNA do fio retardatário, é necessário um complexo de RNA polimerase chamado primase. O primase, que é capaz de unir nucleotídeos de RNA sem requerer uma cadeia pré-existente de ácido nucleico, primeiro adiciona vários nucleotídeos de RNA comlementares em oposição aos nucleotídeos de DNA na cadeia mãe. Isto forma o que é chamado de primer de RNA (veja a Figura \(\PageIndex{10}}).
DNA polimerase III então substitui a primase e é capaz de adicionar nucleotídeos de DNA ao primer de RNA (veja a Figura \PageIndex{11}). Mais tarde, a DNA polimerase II digere o primer de RNA e substitui os nucleotídeos de RNA do primer pelos nucleotídeos de DNA apropriados para preencher a lacuna (ver Figura \PageIndex{12}}). Finalmente, os fragmentos de DNA em si são ligados pela enzima ligase de DNA ligase (veja a Figura 9). No entanto, mesmo com este procedimento complicado, uma macromolécula de 1000 micrómetros de comprimento de ADN bem embalado e super-revestido pode fazer uma cópia exacta de si mesmo em apenas cerca de 10 minutos em condições óptimas, inserindo nucleótidos a uma taxa de cerca de 1000 nucleótidos por segundo!
>br>
br>>div>
br>>Animação de replicação de DNA, #2.
p>Cortesia do HHMI’s Biointeractive. >br>
>br>