Referências >> Primer PCR
Directrizes de desenho do Primer PCR
PCR (Polymerase Chain Reaction)
A Reacção em Cadeia da Polimerase é amplamente considerada uma das mais importantes invenções do século XX em biologia molecular. Pequenas quantidades do material genético podem agora ser amplificadas para ser capaz de identificar, manipular o DNA, detectar organismos infecciosos, incluindo os vírus que causam AIDS, hepatite, tuberculose, detectar variações genéticas, incluindo mutações, em genes humanos e inúmeras outras tarefas.
PCR envolve os três passos seguintes: Desnaturação, recozimento e extensão. Primeiro, o material genético é desnaturado, convertendo as moléculas de DNA duplamente encalhadas em filamentos simples. Os primers são então recozidos para as regiões complementares das moléculas de encalhamento simples. Na terceira etapa, eles são estendidos pela ação da DNA polimerase. Todas estas etapas são sensíveis à temperatura e a escolha comum de temperaturas é 94oC, 60oC e 70oC, respectivamente. Um bom design do primer é essencial para o sucesso das reacções. As importantes considerações de design descritas abaixo são a chave para uma amplificação específica com alto rendimento. Os valores preferidos indicados são incorporados em todos os nossos produtos por defeito.
1. Comprimento do Primário: É geralmente aceite que o comprimento óptimo dos iniciadores de PCR é de 18-22 bp. Este comprimento é suficientemente longo para uma especificidade adequada e suficientemente curto para os iniciadores se ligarem facilmente ao modelo à temperatura de recozimento.
2. Temperatura de fusão dos iniciadores: Temperatura de fusão do primer (Tm) por definição é a temperatura à qual metade do DNA duplex se dissociará para se tornar um encadeamento único e indica a estabilidade do duplex. Primers com temperaturas de fusão na faixa de 52-58 oC geralmente produzem os melhores resultados. Primers com temperaturas de fusão acima de 65oC têm uma tendência para o recozimento secundário. O conteúdo de GC da sequência dá uma indicação justa do primer Tm. Todos os nossos produtos calculam-no utilizando a teoria termodinâmica vizinha mais próxima, aceite como um método muito superior de estimativa, que é considerado o mais recente e melhor disponível.
Fórmula para cálculo do primer Tm:
Temperatura de fusão Tm(K)={ΔH/ ΔS + R ln(C)}, Ou Temperatura de fusão Tm(oC) = {ΔH/ ΔS + R ln(C)} – 273.15 onde
ΔH (kcal/mole) : H é o Enthalpy. Enthalpy é a quantidade de energia térmica possuída pelas substâncias. ΔH é a mudança na Enthalpy. Na fórmula acima a ΔH é obtida pela soma de todos os pares de di-nucleótidos entalpia de cada par de base vizinho mais próximo.
ΔS (kcal/mole) : S é a quantidade de desordem que um sistema apresenta, é chamada entropia. ΔS é a mudança na Entropia. Aqui é obtido pela soma de todos os valores de entropia dos pares de di-nucleotídeos de cada par de base vizinho mais próximo. Uma correção adicional de sal é adicionada uma vez que os parâmetros do vizinho mais próximo foram obtidos a partir de estudos de derretimento de DNA realizados em tampão Na+ 1M e esta é a condição padrão usada para todos os cálculos.
ΔS (correção de sal) = ΔS (1M NaCl )+ 0.368 x N x ln()
Where
N é o número de pares de nucleotídeos no primer ( primer comprimento -1).
é equivalente a sal em mM.
cálculo:
= Concentração de íon monovalente +4 x Mg2+.
3. Temperatura de recozimento do primer: A temperatura de fusão do primer é a estimativa da estabilidade do híbrido DNA-DNA e é crítica para determinar a temperatura de recozimento. Uma Ta demasiado elevada produzirá uma hibridização de primer-template insuficiente resultando num baixo rendimento do produto PCR. Uma Ta demasiado baixa pode possivelmente levar a produtos não específicos causados por um elevado número de desajustes de pares de bases. Verifica-se que a tolerância de não correspondência tem a maior influência na especificidade da PCR.
Ta = 0.3 x Tm(primer) + 0.7 Tm (produto) – 14.9
where,
Tm(primer) = Temperatura de fusão dos primários
Tm(product) = Temperatura de fusão do produto
4. Conteúdo de GC: O conteúdo de GC (o número de G’s e C’s no primário como percentagem do total das bases) do primário deve ser 40-60%.
5. GC Grampo: A presença de bases de G ou C dentro das últimas cinco bases a partir da extremidade de 3′ dos primários (GC clamp) ajuda a promover uma ligação específica na extremidade de 3′ devido à ligação mais forte das bases de G e C. Mais de 3 G’s ou C’s devem ser evitados nas últimas 5 bases do final de 3′ do primer.
6. Estruturas Secundárias Primer: A presença das estruturas secundárias primer produzidas por interações intermoleculares ou intramoleculares pode levar a um fraco ou nenhum rendimento do produto. Elas afetam negativamente o recozimento do primer modelo e, portanto, a amplificação. Reduzem grandemente a disponibilidade de primers para a reacção.
i) Grampos de cabelo: É formado pela interação intramolecular dentro do primer e deve ser evitada. O ideal é um grampo de 3′ com um ΔG de -2 kcal/mol e um grampo interno com um ΔG de -3 kcal/mol é tolerado em geral.
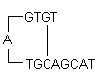
ΔG definição: O Gibbs Free Energy G é a medida da quantidade de trabalho que pode ser extraído de um processo operando a uma pressão constante. É a medida da espontaneidade da reação. A estabilidade do grampo capilar é normalmente representada pelo seu valor ΔG, a energia necessária para quebrar a estrutura secundária. Um valor negativo maior para ΔG indica grampos capilares estáveis e indesejáveis. A presença de grampos capilares na extremidade de 3′ afeta mais adversamente a reação.
ΔG = ΔH – TΔS
ii) Auto-dímero: Um auto-dímero é formado por interações intermoleculares entre os dois (mesmo sentido) primers, onde o primer é homólogo a si mesmo. Geralmente uma grande quantidade de primers é usada em PCR em comparação com a quantidade do gene alvo. Quando os iniciadores formam dímeros intermoleculares muito mais rapidamente do que a hibridação para o ADN alvo, reduzem o rendimento do produto. O ideal é um auto-dímero final de 3′ com um ΔG de -5 kcal/mol e um auto-dímero interno com um ΔG de -6 kcal/mol é geralmente tolerado.
iii) Dímero cruzado: Os primers de dímeros cruzados são formados pela interação intermolecular entre os primers de sentido e os primers antisenso, onde eles são homólogos. Geralmente é tolerado um dímero cruzado de 3′ com um ΔG de -5 kcal/mol e um dímero cruzado interno com um ΔG de -6 kcal/mol.
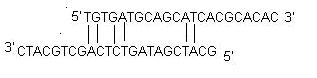
7. Repetindo: Uma repetição é um di-nucleótido que ocorre muitas vezes consecutivas e deve ser evitada porque pode ser mal interpretado. Por exemplo, uma repetição é um di-nucleótido que ocorre muitas vezes consecutivas: ATATATAT. Um número máximo de repetições de di-nucleotídeos aceitáveis em um oligo é 4 di-nucleotídeos.
8. Execuções: Primers com longos tempos de uma única base devem geralmente ser evitados, pois eles podem errar aprimetagem. Por exemplo, AGCGGGGGGGGG tem execuções de base ‘G’ de valor 5 e 4. Um número máximo de execuções aceitas é 4bp.
9. 3′ End Stability (Estabilidade Final): É o valor máximo ΔG das cinco bases a partir do valor 3′ final. Um fim instável de 3′ (menos negativo ΔG) resultará em menos escorva falsa.
10. Evite o modelo de estrutura secundária: Uma única sequência de ácido nucleico é altamente instável e dobra-se em conformações (estruturas secundárias). A estabilidade dessas estruturas secundárias modelo depende muito de sua energia livre e temperaturas de fusão(Tm). A consideração das estruturas secundárias modelo é importante na concepção de primários, especialmente em qPCR. Se os primários forem concebidos em estruturas secundárias estáveis mesmo acima das temperaturas de recozimento, os primários são incapazes de se ligar ao modelo e o rendimento do produto PCR é significativamente afectado. Assim, é importante projetar primers nas regiões dos gabaritos que não formam estruturas secundárias estáveis durante a reação de PCR. Nossos produtos determinam as estruturas secundárias do gabarito e projetam os primers evitando-as.
11. Evite a Homologia Cruzada: Para melhorar a especificidade dos primers é necessário evitar regiões de homologia. Os primers desenhados para uma sequência não devem amplificar outros genes na mistura. Geralmente, os primers são desenhados e depois BLASTED para testar a especificidade. Os nossos produtos oferecem uma alternativa melhor. Você pode evitar regiões de homologia cruzada enquanto desenha os primers. Você pode BLASTAR os modelos contra a base de dados apropriada não redundante e o software irá interpretar os resultados. Ele irá identificar regiões de homologias cruzadas significativas em cada template e evitá-las durante a pesquisa de primers.
Parametros para Design de Pares de Primers
1. Comprimento do Amplicon: O comprimento do amplicon é ditado pelos objetivos experimentais. Para qPCR, o comprimento do alvo é mais próximo de 100 bp e para PCR padrão, é próximo de 500 bp. Se você conhece as posições de cada primer em relação ao modelo, o produto é calculado como: Comprimento do produto = (Posição do primer antisense – Posição do primer de sentido) + 1.
2. Posição do produto: O primer pode ser localizado perto da extremidade de 5′, da extremidade de 3′ ou em qualquer lugar dentro do comprimento especificado. Geralmente, a seqüência próxima ao final de 3′ é conhecida com maior confiança e, portanto, preferida com maior freqüência.
3. Tm de Produto: Temperatura de fusão (Tm) é a temperatura à qual metade do DNA duplex se dissociará e se tornará um único encalhamento. A estabilidade do DNA duplex primer-template pode ser medida pela temperatura de fusão (Tm).
4. Temperatura ótima de recozimento (Ta Opt): A fórmula de Rychlik é a mais respeitada. Nossos produtos usam esta fórmula para calculá-la e milhares de nossos clientes têm relatado bons resultados usando-a para a etapa de recozimento do ciclo PCR. Normalmente resulta num bom rendimento do produto PCR com uma produção mínima de produto falso.
Ta Opt = 0.3 x(Tm de primário) + 0.7 x(Tm de produto) – 14.9
onde
Tm de primer é a temperatura de fusão do par primer-template menos estável
Tm de produto é a temperatura de fusão do produto PCR.
5. Cálculo de Incompatibilidade do Par de Primários Tm: Os dois primers de um par de primers devem ter temperaturas de fusão muito próximas para maximizar o rendimento do produto PCR. A diferença de 5oC ou mais pode não levar a nenhuma amplificação.
Primer Design usando o Software
Está disponível um número de ferramentas de design de iniciadores que podem ajudar no design de iniciadores PCR tanto para utilizadores novos como para utilizadores experientes. Estas ferramentas podem reduzir o custo e tempo envolvidos na experimentação, diminuindo as hipóteses de falha na experimentação.
Primer Premier segue todas as directrizes especificadas para o desenho do primer PCR. Primer Premier pode ser usado para o projeto de primers para modelos únicos, alinhamentos, projeto de primers degenerados, análise enzimática de restrição. análise de contigente e projeto de primers sequenciais.
As diretrizes para o projeto de primers qPCR variam ligeiramente. Softwares como AlleID e Beacon Designer podem projetar primers e sondas de oligonucleotídeos para ensaios de detecção complexos, como ensaios multiplex, design de primers de espécies cruzadas, design de primers específicos para espécies e design de primers para reduzir o custo da experimentação.
PrimerPlex é um software que pode projetar primers para PCR Multiplex e ensaios de genotipagem SNP Multiplex.