B. DNA-Replikation in Bakterien
Im Allgemeinen erfolgt die Replikation der DNA durch Abwickeln der Helix, Trennung der Stränge durch Aufbrechen der Wasserstoffbrücken zwischen den komplementären Strängen und Synthese zweier neuer Stränge durch komplementäre Basenpaarung. Die Replikation beginnt an einer bestimmten Stelle in der DNA, dem Replikationsursprung (oriC).
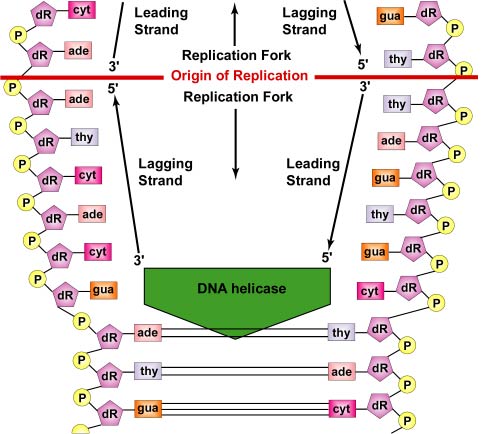
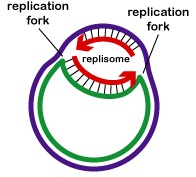
Die DNA-Replikation erfolgt vom Replikationsursprung aus in beide Richtungen. Um die DNA-Replikation zu beginnen, wickeln Enzyme, die so genannten DNA-Helikasen, kurze Abschnitte der beiden übergeordneten DNA-Stränge ab und trennen sie am Replikationsursprung voneinander, um zwei Y-förmige Replikationsgabeln zu bilden. Diese Replikationsgabeln sind der eigentliche Ort der DNA-Kopie (Abbildung \(\PageIndex{3}\)). Alle an der DNA-Replikation beteiligten Proteine sammeln sich an den Replikationsgabeln zu einem Replikationskomplex, dem Replisom (Abbildung \(\PageIndex{4}\)).
Einzelstrangbindende Proteine binden an die einzelsträngigen Bereiche, so dass sich die beiden Stränge nicht wieder verbinden. Durch das Abwickeln der doppelsträngigen Helix entstehen vor der Replikationsgabel positive Supercoils. Enzyme, so genannte Topoisomerasen, wirken dem entgegen, indem sie Brüche in der DNA erzeugen und diese dann wieder zusammenfügen, um negative Supercoils zu bilden und so den Stress im helikalen Molekül während der Replikation zu verringern.
Während sich die Stränge weiter abwickeln und in beide Richtungen um das gesamte DNA-Molekül herum aufspalten, entstehen neue komplementäre Stränge durch Wasserstoffbrückenbindungen zwischen freien DNA-Nukleotiden und denen auf jedem Elternstrang. Während sich die neuen Nukleotide durch Wasserstoffbrückenbindungen gegenüber jedem Elternstrang anordnen, verbinden Enzyme, die so genannten DNA-Polymerasen, die Nukleotide durch Phosphodiesterbindungen. Bei den Nukleotiden, die sich durch komplementäre Basenpaarung aneinanderreihen, handelt es sich eigentlich um Desoxynukleotidtriphosphate, die aus einer stickstoffhaltigen Base, Desoxyribose und drei Phosphaten bestehen. Bei der Bildung der Phosphodiester-Bindung zwischen der 5′-Phosphatgruppe des neuen Nukleotids und dem 3′-OH des letzten Nukleotids im DNA-Strang werden zwei der Phosphate entfernt, wodurch Energie für die Bindung bereitgestellt wird (siehe Abbildung \(\PageIndex{6}\)). Am Ende dient jeder Elternstrang als Vorlage für die Synthese einer komplementären Kopie von sich selbst, so dass zwei identische DNA-Moleküle entstehen (siehe Abbildung \(\PageIndex{7}\)). In Bakterien sorgen die Par-Proteine dafür, dass die bakteriellen Chromosomen während der Zellteilung auf die gegenüberliegenden Pole der Zelle verteilt werden. Sie binden an den Replikationsursprung der DNA und ziehen oder schieben die Chromosomen physisch auseinander, ähnlich wie der mitotische Apparat eukaryontischer Zellen. Fts-Proteine, wie FtsK im Divisom, helfen auch bei der Trennung des replizierten bakteriellen Chromosoms.
GIF-Animation zur Veranschaulichung der DNA-Replikation durch komplementäre Basenpaarung
In Wirklichkeit ist die DNA-Replikation aufgrund der Beschaffenheit der DNA-Polymerasen komplizierter als dies. Die Enzyme der DNA-Polymerase sind nur in der Lage, die Phosphatgruppe am 5′-Kohlenstoff eines neuen Nukleotids mit der Hydroxylgruppe (OH) des 3′-Kohlenstoffs eines bereits in der Kette vorhandenen Nukleotids zu verbinden. Daher kann die DNA nur in 5′- zu 3′-Richtung synthetisiert werden, während ein Elternstrang, der in 3′- zu 5′-Richtung verläuft, kopiert wird.
Jeder DNA-Strang hat zwei Enden. Das 5′-Ende der DNA ist dasjenige mit der endständigen Phosphatgruppe am 5′-Kohlenstoff der Desoxyribose; das 3′-Ende ist dasjenige mit einer endständigen Hydroxylgruppe (OH) an der Desoxyribose des 3′-Kohlenstoffs der Desoxyribose (siehe Abbildung \(\PageIndex{8})). Die beiden Stränge sind antiparallel, d. h. sie verlaufen in entgegengesetzter Richtung. Daher kann ein Elternstrang – der von 3′ nach 5′ verlaufende und als Führungsstrang bezeichnete Strang – direkt über seine gesamte Länge kopiert werden (siehe Abbildung \(\PageIndex{9}\)). Der andere Elternstrang – der von 5′ nach 3′ verlaufende Strang, der so genannte nachlaufende Strang – muss jedoch beim Abwickeln der DNA diskontinuierlich in kurzen Fragmenten (Okazaki-Fragmente) von jeweils etwa 100-1000 Nukleotiden kopiert werden. Dies geschieht, wie bereits erwähnt, am Replisom. Der nachlaufende DNA-Strang schlingt sich vom vorlaufenden Strang ab und ermöglicht es dem Replisom, sich entlang beider Stränge zu bewegen und die DNA bei der Replikation mitzuziehen. Es ist die eigentliche DNA, nicht die DNA-Polymerase, die sich während der bakteriellen DNA-Replikation bewegt (siehe Abbildung \(\PageIndex{5}\)).
Außerdem können die DNA-Polymerase-Enzyme keine neue DNA-Kette von Grund auf beginnen. Sie können nur neue Nukleotide an die 3′-OH-Gruppe eines Nukleotids in einem bereits existierenden Strang anhängen. Um die Synthese des führenden Strangs und jedes DNA-Fragments des nachgeschalteten Strangs zu beginnen, ist daher ein RNA-Polymerase-Komplex, die so genannte Primase, erforderlich. Die Primase, die in der Lage ist, RNA-Nukleotide zu verknüpfen, ohne einen bereits vorhandenen Nukleinsäurestrang zu benötigen, fügt zunächst mehrere ergänzende RNA-Nukleotide gegenüber den DNA-Nukleotiden des Ausgangsstrangs hinzu. Dadurch entsteht ein so genannter RNA-Primer (siehe Abbildung \(\PageIndex{10}\)).
Die DNA-Polymerase III ersetzt dann die Primase und ist in der Lage, DNA-Nukleotide an den RNA-Primer anzuhängen (siehe Abbildung \(\PageIndex{11}\)). Danach verdaut die DNA-Polymerase II den RNA-Primer und ersetzt die RNA-Nukleotide des Primers durch die richtigen DNA-Nukleotide, um die Lücke zu füllen (siehe Abbildung \(\PageIndex{12}\)). Schließlich werden die DNA-Fragmente selbst durch das Enzym DNA-Ligase aneinander gehängt (siehe Abbildung \(\PageIndex{9}\)). Doch selbst mit diesem komplizierten Verfahren kann ein 1000 Mikrometer langes Makromolekül aus dicht gepackter, supergewickelter DNA unter optimalen Bedingungen in nur etwa 10 Minuten eine exakte Kopie von sich selbst herstellen, wobei es Nukleotide mit einer Geschwindigkeit von etwa 1000 Nukleotiden pro Sekunde einfügt!
Animation der DNA-Replikation.
Mit freundlicher Genehmigung von HHMI’s Biointeractive.