B. DNA Replicatie in Bacteriën
In het algemeen wordt DNA gerepliceerd door het afrollen van de helix, het scheiden van de strengen door het verbreken van de waterstofbruggen tussen de complementaire strengen, en de synthese van twee nieuwe strengen door complementaire basenparen. De replicatie begint op een specifieke plaats in het DNA die de oorsprong van de replicatie (oriC) wordt genoemd.
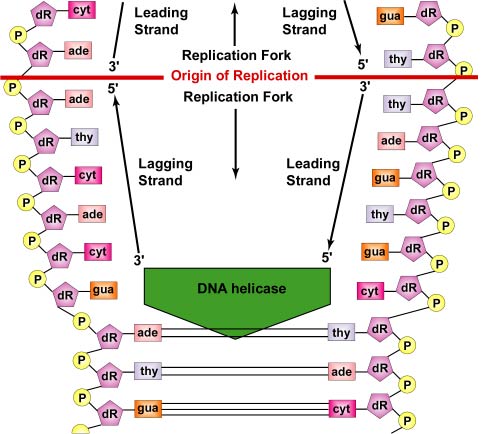
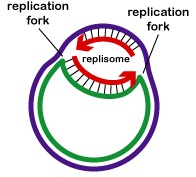
DNA-replicatie verloopt in twee richtingen vanaf de oorsprong van de replicatie. Om met de DNA-replicatie te beginnen, zorgen afwikkelenzymen, DNA-helicasen genaamd, ervoor dat korte segmenten van de twee DNA-ouderstrengen zich bij de replicatiehaard afwikkelen en van elkaar loskomen om twee “Y”-vormige replicatievorken te vormen. Deze replicatievorken zijn de eigenlijke plaats waar het DNA wordt gekopieerd (figuur). Alle eiwitten die betrokken zijn bij de DNA-replicatie worden bij de replicatievorken samengevoegd tot een replicatiecomplex dat replisoom wordt genoemd (figuur
Eenstrengbindende eiwitten binden zich aan de enkelstrengsregio’s zodat de twee strengen niet opnieuw samenkomen. Het afwikkelen van de dubbelstrengshelix genereert positieve supercoils vóór de replicatievork. Enzymen, topoisomerases genaamd, gaan dit tegen door breuken in het DNA te veroorzaken en deze vervolgens weer samen te voegen tot negatieve supercoils om de spanning in het spiraalvormige molecuul tijdens de replicatie te verlichten.
Terwijl de strengen zich blijven afwikkelen en zich in beide richtingen rond de gehele DNA-molecule blijven scheiden, worden nieuwe complementaire strengen geproduceerd door de waterstofbinding van vrije DNA-nucleotiden met die op elke ouderstreng. Terwijl de nieuwe nucleotiden door waterstofbinding tegenover elke ouderstreng komen te liggen, verbinden enzymen, DNA-polymerasen genaamd, de nucleotiden door middel van fosfodiësterbindingen. In feite zijn de nucleotiden die tegenover elkaar liggen door complementaire basenparen deoxynucleotide trifosfaten, bestaande uit een stikstofhoudende base, desoxyribose, en drie fosfaten. Wanneer de fosfodiesterverbinding wordt gevormd tussen de 5′ fosfaatgroep van de nieuwe nucleotide en de 3′ OH van de laatste nucleotide in de DNA-streng, worden twee van de fosfaten verwijderd, waardoor energie vrijkomt voor de binding (zie figuur). Uiteindelijk dient elke ouderstreng als sjabloon om een complementaire kopie van zichzelf te synthetiseren, wat resulteert in de vorming van twee identieke DNA-moleculen (zie figuur \PageIndex{7}). In bacteriën zorgen Par-eiwitten ervoor dat de bacteriële chromosomen tijdens de celdeling naar de tegenoverliggende polen van de cel worden gebracht. Zij binden zich aan de oorsprong van de replicatie van het DNA en trekken of duwen de chromosomen fysiek uit elkaar, vergelijkbaar met het mitotisch apparaat van eukaryotische cellen. Fts-eiwitten, zoals FtsK in het divisoom, helpen ook bij het scheiden van het gerepliceerde bacteriële chromosoom.
GIF-animatie ter illustratie van DNA-replicatie door complementaire basenparen
In werkelijkheid is de DNA-replicatie ingewikkelder dan dit door de aard van de DNA-polymerasen. DNA-polymerase-enzymen zijn alleen in staat om de fosfaatgroep op de 5′ koolstof van een nieuwe nucleotide te verbinden met de hydroxylgroep (OH) van de 3′ koolstof van een nucleotide dat al in de keten zit. Als gevolg daarvan kan DNA alleen worden gesynthetiseerd in een 5′ tot 3′ richting, terwijl een ouderstreng in een 3′ tot 5′ richting wordt gekopieerd.
Elke DNA-streng heeft twee uiteinden. Het 5′-uiteinde van het DNA is dat met de eindfosfaatgroep op het 5′-koolstof van de deoxyribose; het 3′-uiteinde is dat met een eindhydroxylgroep (OH) op het 3′-koolstof van de deoxyribose (zie figuur). De twee strengen zijn antiparallel, dat wil zeggen dat ze in tegengestelde richting lopen. Daarom kan de ene ouderstreng – de streng die van 3′ naar 5′ loopt en de leidende streng wordt genoemd – direct over de gehele lengte worden gekopieerd (zie figuur). De andere ouderstreng – de streng die van 5′ naar 3′ loopt en de achterblijvende streng wordt genoemd – moet echter discontinu in korte fragmenten (Okazaki-fragmenten) van elk ongeveer 100-1000 nucleotiden worden gekopieerd terwijl het DNA zich afwikkelt. Dit gebeurt, zoals hierboven vermeld, in het replisoom. De achterblijvende DNA-streng maakt een lus ten opzichte van de leidende streng en dit stelt het replisoom in staat langs beide strengen te bewegen en het DNA erdoor te trekken terwijl de replicatie plaatsvindt. Het is het eigenlijke DNA en niet het DNA-polymerase dat beweegt tijdens de replicatie van bacterieel DNA (zie figuur).
Dna-polymerase-enzymen kunnen bovendien geen nieuwe DNA-keten vanaf nul beginnen. Zij kunnen alleen nieuwe nucleotiden vastmaken aan de 3′ OH groep van een nucleotide in een reeds bestaande streng. Daarom is voor het starten van de synthese van de voorste streng en elk DNA-fragment van de achterste streng een RNA-polymerasecomplex nodig dat primase wordt genoemd. Het primase, dat in staat is RNA-nucleotiden te verbinden zonder dat daarvoor een reeds bestaande nucleïnezuurstreng nodig is, voegt eerst verscheidene complementaire RNA-nucleotiden toe tegenover de DNA-nucleotiden op de moederstreng. Dit vormt een zogenaamde RNA-primer (zie figuur)
DNA polymerase III vervangt vervolgens de primase en is in staat DNA-nucleotiden aan de RNA-primer toe te voegen (zie figuur)
DNA polymerase III vervangt vervolgens de primase en is in staat DNA-nucleotiden aan de RNA-primer toe te voegen (zie figuur). Later verteert DNA-polymerase II de RNA-primer en vervangt de RNA-nucleotiden van de primer door de juiste DNA-nucleotiden om het gat op te vullen (zie figuur 12). Tenslotte worden de DNA-fragmenten zelf aan elkaar gehaakt door het enzym DNA-ligase (zie figuur). Maar zelfs met deze gecompliceerde procedure kan een 1000 micrometer lange macromolecule van strak opeengepakt, supergewikkeld DNA een exacte kopie van zichzelf maken in slechts ongeveer 10 minuten tijd onder optimale omstandigheden, waarbij nucleotiden worden ingebracht met een snelheid van ongeveer 1000 nucleotiden per seconde!
Animatie van DNA replicatie.
Courtesy of HHMI’s Biointeractive.