Referenties >> PCR Primer Ontwerp Richtlijnen
PCR (Polymerase Chain Reaction)
Polymerase Chain Reaction wordt algemeen beschouwd als een van de belangrijkste uitvindingen van de 20e eeuw in de moleculaire biologie. Kleine hoeveelheden genetisch materiaal kunnen nu worden geamplificeerd om DNA te identificeren, te manipuleren, besmettelijke organismen op te sporen, waaronder de virussen die AIDS, hepatitis en tuberculose veroorzaken, genetische variaties, waaronder mutaties, in menselijke genen op te sporen en tal van andere taken uit te voeren.
PCR omvat de volgende drie stappen: Denaturatie, gloeien en verlenging. Eerst wordt het genetisch materiaal gedenatureerd, waarbij de dubbelstrengs DNA-moleculen worden omgezet in enkelstrengs. De primers worden vervolgens geannealed op de complementaire gebieden van de enkelstrengs moleculen. In de derde stap worden ze verlengd door de werking van het DNA-polymerase. Al deze stappen zijn temperatuurgevoelig en de gangbare temperatuurkeuze is respectievelijk 94oC, 60oC en 70oC. Een goed primerontwerp is essentieel voor succesvolle reacties. De hieronder beschreven belangrijke ontwerpoverwegingen vormen de sleutel tot een specifieke amplificatie met een hoge opbrengst. De aangegeven voorkeurswaarden zijn standaard in al onze producten ingebouwd.
1. Lengte van de primer: Algemeen wordt aangenomen dat de optimale lengte van PCR-primers 18-22 bp is. Deze lengte is lang genoeg voor voldoende specificiteit en kort genoeg voor primers om zich bij de annealingtemperatuur gemakkelijk aan het sjabloon te binden.
2. Smelttemperatuur van de primer: Primer Melting Temperature (Tm) is per definitie de temperatuur waarbij de ene helft van het DNA-duplex zal dissociëren om enkelstrengs te worden en geeft de duplexstabiliteit aan. Primers met een smelttemperatuur tussen 52 en 58 oC geven over het algemeen de beste resultaten. Primers met smelttemperaturen boven 65oC hebben de neiging tot secundaire annealing. Het GC-gehalte van de sequentie geeft een goede indicatie van de primer Tm. Al onze producten berekenen het met behulp van de nearest neighbor thermodynamische theorie, aanvaard als een veel betere methode voor het schatten ervan, die wordt beschouwd als de meest recente en beste beschikbare.
Formule voor primer Tm berekening:
Smelttemperatuur Tm(K)={ΔH/ ΔS + R ln(C)}, Of Smelttemperatuur Tm(oC) = {ΔH/ ΔS + R ln(C)} – 273,15 waarin
ΔH (kcal/mole) : H is de Enthalpie. Enthalpie is de hoeveelheid warmte-energie die een stof bezit. ΔH is de verandering in Enthalpie. In de bovenstaande formule wordt de ΔH verkregen door alle enthalpiewaarden van de di-nucleotideparen van elk naburig basenpaar bij elkaar op te tellen.
ΔS (kcal/mole) : S is de mate van wanorde die een systeem vertoont en wordt entropie genoemd. ΔS is de verandering in entropie. Hier wordt deze verkregen door alle entropiewaarden van de di-nucleotideparen van elk naburig basenpaar bij elkaar op te tellen. Er wordt een extra zoutcorrectie toegevoegd aangezien de Nearest Neighbor-parameters werden verkregen uit DNA-smeltingsstudies die werden uitgevoerd in 1M Na+ buffer en dit is de standaardvoorwaarde die voor alle berekeningen wordt gebruikt.
ΔS (zoutcorrectie) = ΔS (1M NaCl )+ 0.368 x N x ln()
Waar
N het aantal nucleotideparen in de primer is ( primerlengte -1).
is het zoutequivalent in mM.
berekening:
= Monovalente ionenconcentratie +4 x vrij Mg2+.
3. Primer-afsmelttemperatuur: De smelttemperatuur van de primer is de schatting van de stabiliteit van de DNA-DNA-hybride en doorslaggevend voor het bepalen van de annealingtemperatuur. Een te hoge Ta leidt tot onvoldoende primer-template hybridisatie, wat resulteert in een lage PCR-productopbrengst. Een te lage Ta kan mogelijk tot niet-specifieke producten leiden als gevolg van een groot aantal basenpaarmismatches. Mismatch tolerantie blijkt de sterkste invloed te hebben op PCR specificiteit.
Ta = 0.3 x Tm(primer) + 0.7 Tm (product) – 14.9
waar,
Tm(primer) = smelttemperatuur van de primers
Tm(product) = smelttemperatuur van het product
4. GC-gehalte: Het GC-gehalte (het aantal G’s en C’s in de primer als percentage van het totaal aantal basen) van de primer moet 40-60% zijn.
5. GC-klem: De aanwezigheid van G- of C-basen binnen de laatste vijf basen vanaf het 3′-uiteinde van primers (GC-klem) helpt specifieke binding aan het 3′-uiteinde te bevorderen door de sterkere binding van G- en C-basen. Meer dan 3 G’s of C’s moeten worden vermeden in de laatste 5 basen aan het 3′-uiteinde van de primer.
6. Primer Secundaire Structuren: Aanwezigheid van de primer secundaire structuren geproduceerd door intermoleculaire of intramoleculaire interacties kan leiden tot slechte of geen opbrengst van het product. Zij hebben een ongunstig effect op de annealing van de primersjabloon en dus op de amplificatie. Zij verminderen in sterke mate de beschikbaarheid van primers voor de reactie.
i) Haarspelden: Deze worden gevormd door intramoleculaire interactie binnen de primer en moeten worden vermeden. Optimaal is een haarspeld aan het 3′-uiteinde met een ΔG van -2 kcal/mol en een haarspeld aan de binnenkant met een ΔG van -3 kcal/mol, die in het algemeen wordt getolereerd.
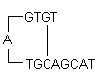
ΔG-definitie: De vrije energie G van Gibbs is de maat voor de hoeveelheid arbeid die kan worden onttrokken aan een proces dat bij een constante druk werkt. Het is de maat voor de spontaniteit van de reactie. De stabiliteit van een haarspeld wordt gewoonlijk weergegeven door zijn ΔG-waarde, de energie die nodig is om de secundaire structuur te breken. Een grotere negatieve waarde voor ΔG wijst op stabiele, ongewenste haarspelden. De aanwezigheid van haarspelden aan het 3′-uiteinde beïnvloedt de reactie het meest ongunstig.
ΔG = ΔH – TΔS
ii) Zelfdimeer: Een primer-zelfdimeer wordt gevormd door intermoleculaire interacties tussen twee primers (met dezelfde zin), waarbij de primer homoloog is aan zichzelf. Over het algemeen wordt bij PCR een grote hoeveelheid primers gebruikt in vergelijking met de hoeveelheid doelgen. Wanneer primers veel gemakkelijker intermoleculaire dimeren vormen dan zich aan het doel-DNA te hybridiseren, verminderen zij de opbrengst van het product. Optimaal is een 3′-eind-zelfdimeer met een ΔG van -5 kcal/mol en een interne zelfdimeer met een ΔG van -6 kcal/mol.
iii) Kruisendimeer: Kruisendimeren van primers worden gevormd door intermoleculaire interactie tussen sense en antisense primers, wanneer deze homoloog zijn. Optimaal is een 3′-uiteinde kruisendimeer met een ΔG van -5 kcal/mol en een interne kruisendimeer met een ΔG van -6 kcal/mol, die over het algemeen worden getolereerd.
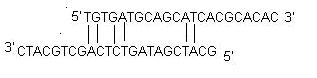
7. Herhalingen: Een herhaling is een di-nucleotide dat vele malen achter elkaar voorkomt en moet worden vermeden omdat ze verkeerd kunnen priemmen. Bijvoorbeeld: ATATATAT. Het maximumaantal di-nucleotide herhalingen in een oligo is 4 di-nucleotiden.
8. Runs: Primers met lange runs van een enkele base moeten in het algemeen worden vermeden, omdat ze kunnen misprimen. AGCGGGATGGGGGG heeft bijvoorbeeld reeksen van de basis “G” van waarde 5 en 4. Het maximumaantal runs dat wordt geaccepteerd is 4bp.
9. 3′ Eindstabiliteit: Dit is de maximale ΔG-waarde van de vijf basen van het 3′-uiteinde. Een onstabiel 3′-uiteinde (minder negatieve ΔG) leidt tot minder valse priming.
10. Vermijd secundaire structuur van het sjabloon: Een enkelstrengs Nucleïnezuursequentie is zeer instabiel en vouwt zich in conformatie (secundaire structuren). De stabiliteit van deze secundaire sjabloonstructuren hangt grotendeels af van hun vrije energie en smelttemperatuur (Tm). Rekening houden met secundaire structuren van het sjabloon is belangrijk bij het ontwerpen van primers, vooral bij qPCR. Als primers worden ontworpen op een secundaire structuur die zelfs boven de annealingtemperaturen stabiel is, kunnen de primers zich niet aan het sjabloon binden en wordt de opbrengst van het PCR-product aanzienlijk beïnvloed. Daarom is het belangrijk primers te ontwerpen in de gebieden van de templates die tijdens de PCR-reactie geen stabiele secundaire structuren vormen. Onze producten bepalen de secundaire structuren van het sjabloon en ontwerpen primers die deze vermijden.
11. Vermijd kruishomologie: Om de specificiteit van de primers te verbeteren is het noodzakelijk om regio’s van homologie te vermijden. Primers die voor een sequentie zijn ontworpen, mogen geen andere genen in het mengsel amplificeren. Gewoonlijk worden primers ontworpen en vervolgens BLASTed om de specificiteit te testen. Onze producten bieden een beter alternatief. U kunt gebieden met kruishomologie vermijden bij het ontwerpen van primers. U kunt de templates BLASTen tegen de juiste niet-redundante database en de software zal de resultaten interpreteren. Het zal regio’s identificeren met significante cross homologies in elk sjabloon en deze vermijden tijdens het primer zoeken.
Parameters voor Primer Pair Ontwerp
1. Ampliconlengte: De lengte van het amplicon wordt bepaald door de experimentele doelen. Voor qPCR ligt de streeflengte dichter bij 100 bp en voor standaard-PCR in de buurt van 500 bp. Als u de posities van elke primer ten opzichte van het sjabloon kent, wordt het product als volgt berekend Productlengte = (Positie van de antisense primer-Positie van de sense primer) + 1.
2. Productpositie: De primer kan zich in de buurt van het 5′-uiteinde, het 3′-uiteinde of waar dan ook binnen de gespecificeerde lengte bevinden. In het algemeen is de sequentie dicht bij het 3′-uiteinde met grotere betrouwbaarheid bekend en verdient daarom de voorkeur.
3. Tm van het product: De smelttemperatuur (Tm) is de temperatuur waarbij de ene helft van het DNA-duplex zal dissociëren en éénstrengs zal worden. De stabiliteit van de primer-template DNA duplex kan worden gemeten aan de hand van de smelttemperatuur (Tm).
4. Optimale onthardingstemperatuur (Ta Opt): De formule van Rychlik wordt het meest gerespecteerd. Onze producten gebruiken deze formule voor de berekening en duizenden van onze klanten hebben goede resultaten gemeld bij het gebruik ervan voor de annealingstap van de PCR-cyclus. Het resulteert meestal in een goede PCR-productopbrengst met een minimale productie van vals product.
Ta Opt = 0.3 x(Tm van primer) + 0.7 x(Tm van product) – 14.9
waar
Tm van primer is de smelttemperatuur van het minder stabiele primer-templaatpaar
Tm van product is de smelttemperatuur van het PCR-product.
5. Berekening van de Tm-mismatch van het primerpaar: De twee primers van een primerpaar moeten nauw op elkaar aansluitende smelttemperaturen hebben voor een maximale opbrengst van het PCR-product. Een verschil van 5oC of meer kan leiden tot geen amplificatie.
Primerontwerp met behulp van software
Er zijn een aantal primerontwerphulpmiddelen beschikbaar die zowel nieuwe als ervaren gebruikers kunnen helpen bij het ontwerpen van PCR-primers. Deze tools kunnen de kosten en tijd die gemoeid zijn met experimenten verminderen door de kans op mislukte experimenten te verkleinen.
Primer Premier volgt alle richtlijnen die zijn gespecificeerd voor het ontwerpen van PCR-primers. Primer Premier kan worden gebruikt voor het ontwerpen van primers voor single templates, alignments, degenerate primer design, restrictie enzymen analyse. contig analyse en het ontwerpen van sequencing primers.
De richtlijnen voor qPCR primer design variëren enigszins. Software zoals AlleleID en Beacon Designer kunnen primers en oligonucleotide probes ontwerpen voor complexe detectie-assays zoals multiplex assays, cross species primer design, species specific primer design en primer design om de kosten van experimenten te verlagen.
PrimerPlex is een software die primers kan ontwerpen voor Multiplex PCR en multiplex SNP genotyping assays.