B. Replikacja DNA u bakterii
Ogólnie, DNA replikuje się przez rozwijanie helisy, rozdzielanie nici przez zerwanie wiązań wodorowych między komplementarnymi nićmi i syntezę dwóch nowych nici przez komplementarne parowanie zasad. Replikacja rozpoczyna się w określonym miejscu w DNA zwanym początkiem replikacji (oriC).
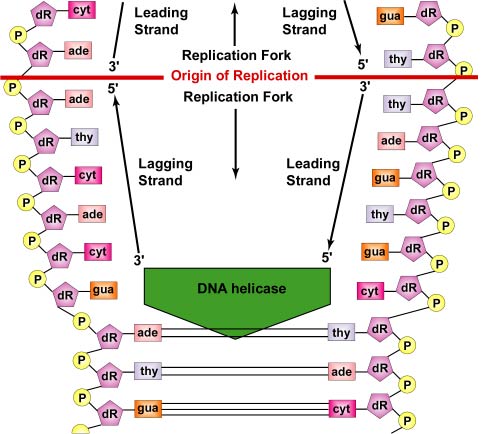
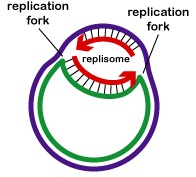
Replikacja DNA jest dwukierunkowa od początku replikacji. Aby rozpocząć replikację DNA, enzymy odwijające zwane helikazami DNA powodują, że krótkie segmenty dwóch macierzystych nici DNA odwijają się i oddzielają od siebie w miejscu początku replikacji, tworząc dwa „Y” w kształcie widełek replikacyjnych. Te widełki replikacyjne są rzeczywistym miejscem kopiowania DNA (rysunek). Wszystkie białka biorące udział w replikacji DNA gromadzą się w widełkach replikacyjnych, tworząc kompleks replikacyjny zwany replikonem (Rysunek \(\)).
Białka wiążące pojedynczą nić wiążą się z regionami jednoniciowymi, więc dwie nici nie łączą się ponownie. Odwijanie dwuniciowej helisy powoduje powstawanie dodatnich superkolejek przed widełkami replikacyjnymi. Enzymy zwane topoizomerazami przeciwdziałają temu, wytwarzając przerwy w DNA, a następnie łączą je, tworząc negatywne superwłókna w celu złagodzenia naprężeń w cząsteczce helisy podczas replikacji.
Jak nici nadal się rozwijają i rozdzielają w obu kierunkach wokół całej cząsteczki DNA, nowe komplementarne nici są wytwarzane przez wiązanie wodorowe wolnych nukleotydów DNA z tymi na każdej nici rodzicielskiej. W miarę jak nowe nukleotydy ustawiają się naprzeciwko każdej nici macierzystej dzięki wiązaniu wodorowemu, enzymy zwane polimerazami DNA łączą nukleotydy za pomocą wiązań fosfodiestrowych. W rzeczywistości nukleotydy łączące się poprzez komplementarne parowanie zasad są trifosforanami deoksynukleotydów, składającymi się z zasady azotowej, deoksyrybozy i trzech fosforanów. Podczas tworzenia wiązania fosfodiestrowego pomiędzy 5′ grupą fosforanową nowego nukleotydu a 3′ OH ostatniego nukleotydu w nici DNA, dwa z fosforanów są usuwane, dostarczając energii do wiązania (patrz rysunek). W końcu każda nić macierzysta służy jako szablon do syntezy komplementarnej kopii samej siebie, w wyniku czego powstają dwie identyczne cząsteczki DNA (patrz rysunek \(\PageIndex{7}}). W bakteriach, białka Par działają w celu oddzielenia chromosomów bakteryjnych do przeciwległych biegunów komórki podczas podziału komórki. Wiążą się one z początkiem replikacji DNA i fizycznie odciągają lub odpychają chromosomy, podobnie jak aparat mitotyczny komórek eukariotycznych. Białka Fts, takie jak FtsK w divisomie, również pomagają w rozdzielaniu zreplikowanego chromosomu bakteryjnego.
Animacja GIF ilustrująca replikację DNA przez komplementarne parowanie zasad
W rzeczywistości replikacja DNA jest bardziej skomplikowana ze względu na naturę polimeraz DNA. Enzymy polimerazy DNA są w stanie jedynie przyłączyć grupę fosforanową przy węglu 5′ nowego nukleotydu do grupy hydroksylowej (OH) przy węglu 3′ nukleotydu już znajdującego się w łańcuchu. W rezultacie DNA może być syntetyzowane tylko w kierunku od 5′ do 3′ podczas kopiowania nici macierzystej biegnącej w kierunku od 3′ do 5′.
Każda nić DNA ma dwa końce. Koniec 5′ DNA to ten z końcową grupą fosforanową na 5′ węglu deoksyrybozy; koniec 3′ to ten z końcową grupą hydroksylową (OH) na 3′ węglu deoksyrybozy (patrz rysunek). Te dwie nici są antyrównoległe, czyli biegną w przeciwnych kierunkach. Dlatego jedna nić macierzysta – ta biegnąca od 3′ do 5′ i zwana wiodącą – może być kopiowana bezpośrednio na całej długości (patrz rysunek). Jednakże druga nić macierzysta – ta biegnąca od 5′ do 3′ i zwana nicią opóźniającą – musi być kopiowana nieciągle w krótkich fragmentach (fragmenty Okazaki) po około 100-1000 nukleotydów każdy w miarę rozwijania się DNA. Dzieje się to, jak wspomniano wyżej, w replikonie. Otaczająca nić DNA odwija się od nici wiodącej, co umożliwia repliksomowi poruszanie się wzdłuż obu nici, przeciągając DNA w trakcie replikacji. To rzeczywiste DNA, a nie polimeraza DNA, porusza się podczas replikacji bakteryjnego DNA (patrz rysunek).
Dodatkowo, enzymy polimerazy DNA nie mogą rozpocząć nowego łańcucha DNA od zera. Mogą jedynie przyłączać nowe nukleotydy do grupy 3′ OH nukleotydu w istniejącej wcześniej nici. Dlatego do rozpoczęcia syntezy nici wiodącej i każdego fragmentu DNA nici opóźniającej potrzebny jest kompleks polimerazy RNA zwany primazą. Primaza, która jest zdolna do łączenia nukleotydów RNA bez konieczności posiadania wcześniej istniejącej nici kwasu nukleinowego, najpierw dodaje kilka komplementarnych nukleotydów RNA naprzeciwko nukleotydów DNA na nici macierzystej. W ten sposób powstaje coś, co nazywa się primerem RNA (patrz rysunek).
Polimeraza DNA III zastępuje primazę i jest w stanie dodać nukleotydy DNA do primera RNA (patrz rysunek). Później polimeraza DNA II trawi primer RNA i zastępuje nukleotydy RNA primera odpowiednimi nukleotydami DNA, aby wypełnić lukę (patrz rysunek \(\PageIndex{12}}). Wreszcie, same fragmenty DNA są łączone ze sobą przez enzym ligazę DNA (patrz rysunek \(\PageIndex{9}\). Jednak nawet przy tak skomplikowanej procedurze, 1000-mikrometrowa makrocząsteczka ciasno upakowanego, superzwiniętego DNA może w optymalnych warunkach wykonać dokładną kopię samej siebie w ciągu zaledwie około 10 minut, wstawiając nukleotydy w tempie około 1000 nukleotydów na sekundę!
Animacja replikacji DNA.
Courtesy of HHMI’s Biointeractive.