Referencje >> PCR Primer
PCR Primer Design Guidelines
PCR (Polymerase Chain Reaction)
Reakcja łańcuchowa polimerazy jest powszechnie uważana za jeden z najważniejszych wynalazków XX wieku w biologii molekularnej. Małe ilości materiału genetycznego mogą być teraz amplifikowane w celu identyfikacji, manipulacji DNA, wykrywania organizmów zakaźnych, w tym wirusów wywołujących AIDS, zapalenie wątroby, gruźlicę, wykrywania zmian genetycznych, w tym mutacji, w ludzkich genach i wielu innych zadań.
PCR obejmuje następujące trzy etapy: Denaturacji, Wyżarzania i Wydłużania. Najpierw materiał genetyczny jest denaturowany, co powoduje przekształcenie dwuniciowych cząsteczek DNA w pojedyncze nici. Startery są następnie wyżarzane do komplementarnych regionów jednoniciowych cząsteczek. W trzecim etapie są one przedłużane przez działanie polimerazy DNA. Wszystkie te etapy są wrażliwe na temperaturę, a powszechnie wybierane temperatury to odpowiednio 94oC, 60oC i 70oC. Dobre zaprojektowanie primera jest niezbędne dla udanych reakcji. Ważne względy dotyczące projektowania opisane poniżej są kluczem do specyficznej amplifikacji z wysoką wydajnością. Wskazane preferowane wartości są domyślnie wbudowane we wszystkie nasze produkty.
1. Długość primera: Ogólnie przyjmuje się, że optymalna długość starterów PCR wynosi 18-22 bp. Długość ta jest wystarczająco długa, aby zapewnić odpowiednią specyficzność i wystarczająco krótka, aby primery łatwo wiązały się z szablonem w temperaturze annealingu.
2. Temperatura topnienia primera: Temperatura topnienia primera (Tm) z definicji jest temperaturą, w której jedna połowa dupleksu DNA ulegnie dysocjacji i stanie się jednoniciowa, co wskazuje na stabilność dupleksu. Primery o temperaturach topnienia w zakresie 52-58 oC generalnie dają najlepsze wyniki. Primery o temperaturach topnienia powyżej 65oC mają tendencję do wtórnego annealingu. Zawartość GC w sekwencji pozwala na dokładną ocenę Tm primera. Wszystkie nasze produkty obliczają ją przy użyciu teorii termodynamicznej najbliższego sąsiedztwa, uznawanej za znacznie lepszą metodę jej szacowania, która jest uważana za najnowszą i najlepszą z dostępnych.
Formuła do obliczania Tm primera:
Temperatura topnienia Tm(K)={ΔH/ ΔS + R ln(C)}, lub Temperatura topnienia Tm(oC) = {ΔH/ ΔS + R ln(C)} – 273.15 gdzie
ΔH (kcal/mol) : H to entalpia. Entalpia to ilość energii cieplnej posiadanej przez substancje. ΔH to zmiana entalpii. W powyższym wzorze ΔH uzyskuje się przez zsumowanie wartości entalpii wszystkich par dwunukleotydowych każdej najbliższej sąsiedniej pary zasad.
ΔS (kcal/mol) : S to ilość nieuporządkowania, jaką wykazuje system, nazywana entropią. ΔS to zmiana w entropii. Tutaj uzyskuje się ją przez zsumowanie wartości entropii wszystkich par dwunukleotydowych każdej najbliższej sąsiedniej pary zasad. Dodatkowa poprawka na sól jest dodawana, ponieważ parametry najbliższego sąsiedztwa zostały uzyskane z badań topnienia DNA przeprowadzonych w buforze 1M Na+ i jest to domyślny warunek używany do wszystkich obliczeń.
ΔS (poprawka na sól) = ΔS (1M NaCl )+ 0.368 x N x ln()
Gdzie
N jest liczbą par nukleotydów w starterze ( długość startera -1).
jest równoważnikiem soli w mM.
Obliczenia:
= stężenie jonów monowalentnych +4 x wolny Mg2+.
3. Temperatura wyżarzania primera: Temperatura topnienia primera jest oszacowaniem stabilności hybrydy DNA-DNA i jest krytyczna przy określaniu temperatury annealingu. Zbyt wysoka Ta spowoduje niewystarczającą hybrydyzację primera z matrycą, co skutkuje niską wydajnością produktu PCR. Zbyt niska Ta może prowadzić do powstawania niespecyficznych produktów spowodowanych dużą liczbą niedopasowań par zasad. Stwierdzono, że tolerancja niedopasowania ma najsilniejszy wpływ na specyficzność PCR.
Ta = 0.3 x Tm(primer) + 0.7 Tm (produkt) – 14.9
gdzie,
Tm(primer) = Temperatura topnienia primerów
Tm(produkt) = Temperatura topnienia produktu
4. Zawartość GC: Zawartość GC (liczba G i C w primerze jako procent całkowitej liczby zasad) w primerze powinna wynosić 40-60%.
5. Zacisk GC: Obecność zasad G lub C w obrębie ostatnich pięciu zasad od 3′ końca primera (zacisk GC) pomaga promować specyficzne wiązanie na 3′ końcu ze względu na silniejsze wiązanie zasad G i C. Należy unikać więcej niż 3 zasad G lub C w ostatnich 5 bazach na 3′ końcu primera.
6. Struktury drugorzędowe primera: Obecność drugorzędowych struktur primera wytwarzanych przez interakcje międzycząsteczkowe lub wewnątrzcząsteczkowe może prowadzić do słabej wydajności produktu lub jej braku. Niekorzystnie wpływają one na wyżarzanie matrycy primera, a tym samym na amplifikację. Znacznie zmniejszają dostępność starterów do reakcji.
i) Spinki do włosów: Powstają w wyniku interakcji wewnątrzcząsteczkowych w obrębie primera i należy ich unikać. Optymalnie 3′ końcowa spinka do włosów o ΔG równym -2 kcal/mol i wewnętrzna spinka do włosów o ΔG równym -3 kcal/mol są ogólnie tolerowane.
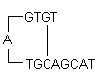
Definicja ΔG: Energia swobodna Gibbsa G jest miarą ilości pracy, jaką można wydobyć z procesu działającego przy stałym ciśnieniu. Jest miarą spontaniczności reakcji. Stabilność spinki do włosów jest powszechnie reprezentowana przez jej wartość ΔG, energię potrzebną do przerwania struktury drugorzędowej. Większa ujemna wartość ΔG wskazuje na stabilne, niepożądane spinki do włosów. Obecność spinek do włosów na końcu 3′ najbardziej negatywnie wpływa na reakcję.
ΔG = ΔH – TΔS
ii) Samodzielny dimer: Samodzielny dimer primera powstaje w wyniku oddziaływań międzycząsteczkowych pomiędzy dwoma (o tym samym znaczeniu) primerami, gdzie primer jest homologiczny do siebie samego. Generalnie w PCR używa się dużej ilości primerów w porównaniu do ilości genu docelowego. Gdy primery tworzą dimery międzycząsteczkowe znacznie łatwiej niż hybrydyzują do docelowego DNA, zmniejszają one wydajność produktu. Optymalnie 3′ końcowy własny dimer o ΔG równym -5 kcal/mol i wewnętrzny własny dimer o ΔG równym -6 kcal/mol są ogólnie tolerowane.
iii) Dimery krzyżowe: Dimery krzyżowe primerów powstają w wyniku interakcji międzycząsteczkowych pomiędzy primerami sensu i antysensu, gdy są one homologiczne. Optymalnie 3′ końcowy dimer krzyżowy z ΔG wynoszącym -5 kcal/mol i wewnętrzny dimer krzyżowy z ΔG wynoszącym -6 kcal/mol jest ogólnie tolerowany.
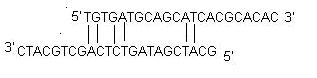
7) Powtórzenia: Powtórzenie jest di-nukleotydem występującym wiele razy po sobie i powinno się go unikać, ponieważ mogą się one błędnie łączyć. Na przykład: ATATATATAT. Maksymalna liczba powtórzeń di-nukleotydów dopuszczalna w oligo to 4 di-nukleotydy.
8. Przebiegi: Podkłady z długimi przebiegami pojedynczej zasady powinny być generalnie unikane, ponieważ mogą się źle aplikować. Na przykład, AGCGGGGATGGGGG ma przebiegi bazy 'G’ o wartości 5 i 4. Maksymalna liczba akceptowanych przebiegów wynosi 4bp.
9. 3′ Stabilność końcowa: Jest to maksymalna wartość ΔG pięciu zasad z końca 3′. Niestabilny koniec 3′ (mniej ujemne ΔG) spowoduje mniej fałszywych primingów.
10. Unikanie Wtórnej Struktury Szablonu: Jednowłóknowe sekwencje kwasu nukleinowego są wysoce niestabilne i składają się w konformacje (struktury drugorzędowe). Stabilność tych struktur wtórnych szablonu zależy w dużej mierze od ich energii swobodnej i temperatury topnienia (Tm). Uwzględnienie struktur drugorzędowych szablonu jest ważne przy projektowaniu starterów, szczególnie w qPCR. Jeśli startery są zaprojektowane na struktury drugorzędowe, które są stabilne nawet powyżej temperatury annealingu, startery nie są w stanie związać się z szablonem i wydajność produktu PCR jest znacznie ograniczona. Dlatego ważne jest, aby projektować primery w regionach szablonów, które nie tworzą stabilnych struktur drugorzędowych podczas reakcji PCR. Nasze produkty określają struktury drugorzędowe szablonu i projektują primery omijające je.
11. Unikanie homologii krzyżowej: Aby poprawić specyficzność starterów konieczne jest unikanie regionów homologii. Primery zaprojektowane dla danej sekwencji nie mogą amplifikować innych genów w mieszaninie. Zazwyczaj startery są projektowane, a następnie sprawdzane metodą BLAST w celu zbadania specyficzności. Nasze produkty oferują lepszą alternatywę. Można uniknąć regionów krzyżowej homologii podczas projektowania primerów. Możesz wykonać BLAST szablonów w stosunku do odpowiedniej nieredundantnej bazy danych, a oprogramowanie zinterpretuje wyniki. Zidentyfikuje znaczące regiony homologii krzyżowej w każdym z szablonów i uniknie ich podczas wyszukiwania primerów.
Parametry do projektowania par primerów
1. Długość amplikonu: Długość amplikonu jest dyktowana przez cele eksperymentu. Dla qPCR, docelowa długość jest bliższa 100 bp, a dla standardowego PCR, jest bliska 500 bp. Jeśli znamy pozycje każdego primera w stosunku do szablonu, produkt jest obliczany jako: Długość produktu = (Position of antisense primer-Position of sense primer) + 1.
2. Product Position: Primer może być umieszczony w pobliżu 5′ końca, 3′ końca lub w dowolnym miejscu w obrębie określonej długości. Generalnie, sekwencja w pobliżu 3′ końca jest znana z większą pewnością i stąd najczęściej preferowana.
3. Tm produktu: Temperatura topnienia (Tm) jest temperaturą, w której jedna połowa dupleksu DNA zdysocjuje i stanie się jednoniciowa. Stabilność dupleksu primer-templat DNA może być mierzona przez temperaturę topnienia (Tm).
4. Optymalna temperatura wyżarzania (Ta Opt): Wzór Rychlika jest najbardziej respektowany. Nasze produkty wykorzystują tę formułę do jej obliczania i tysiące naszych klientów odnotowało dobre wyniki stosując ją do etapu wyżarzania w cyklu PCR. Zazwyczaj daje to dobrą wydajność produktu PCR przy minimalnej produkcji fałszywych produktów.
Ta Opt = 0.3 x(Tm primera) + 0.7 x(Tm produktu) – 14.9
gdzie
Tm primera jest temperaturą topnienia mniej stabilnej pary primer-templat
Tm produktu jest temperaturą topnienia produktu PCR.
5. Obliczanie niedopasowania Tm pary primerów: Dwa primery z pary primerów powinny mieć ściśle dopasowane temperatury topnienia dla maksymalizacji wydajności produktu PCR. Różnica 5oC lub więcej może prowadzić do braku amplifikacji.
Projektowanie primerów przy użyciu oprogramowania
Dostępnych jest wiele narzędzi do projektowania primerów, które mogą pomóc w projektowaniu primerów PCR zarówno dla nowych jak i doświadczonych użytkowników. Narzędzia te mogą zredukować koszty i czas związany z eksperymentowaniem, zmniejszając szanse na nieudane eksperymenty.
Primer Premier stosuje się do wszystkich wytycznych określonych dla projektowania primerów PCR. Primer Premier może być używany do projektowania starterów dla pojedynczych szablonów, wyrównywania, projektowania starterów zdegenerowanych, analizy enzymów restrykcyjnych, analizy kontigów i projektowania starterów sekwencjonujących.
Wytyczne dla projektowania starterów qPCR różnią się nieznacznie. Oprogramowanie takie jak AlleID i Beacon Designer pozwala na projektowanie primerów i sond oligonukleotydowych dla złożonych testów detekcji, takich jak testy multipleksowe, projektowanie primerów dla różnych gatunków, projektowanie primerów specyficznych dla danego gatunku oraz projektowanie primerów w celu zmniejszenia kosztów eksperymentów.
PrimerPlex jest oprogramowaniem, które może projektować primery dla testów Multiplex PCR i multipleksowych testów genotypowania SNP.