Referenzen >> PCR Primer
PCR Primer Design Guidelines
PCR (Polymerase Chain Reaction)
Die Polymerase Chain Reaction gilt weithin als eine der wichtigsten Erfindungen des 20. Jahrhunderts in der Molekularbiologie. Jahrhunderts in der Molekularbiologie. Kleine Mengen des genetischen Materials können nun vervielfältigt werden, um DNA zu identifizieren, zu manipulieren, infektiöse Organismen nachzuweisen, einschließlich der Viren, die AIDS, Hepatitis und Tuberkulose verursachen, genetische Variationen, einschließlich Mutationen, in menschlichen Genen nachzuweisen und zahlreiche andere Aufgaben zu erfüllen.
Die PCR umfasst die folgenden drei Schritte: Denaturierung, Annealing und Extension. Zunächst wird das genetische Material denaturiert, wobei die doppelsträngigen DNA-Moleküle in Einzelstränge umgewandelt werden. Anschließend werden die Primer an die komplementären Bereiche der einzelsträngigen Moleküle angelagert. Im dritten Schritt werden sie durch die Wirkung der DNA-Polymerase verlängert. Alle diese Schritte sind temperaturabhängig, und die übliche Wahl der Temperaturen ist 94oC, 60oC bzw. 70oC. Ein gutes Primerdesign ist für erfolgreiche Reaktionen unerlässlich. Die im Folgenden beschriebenen wichtigen Überlegungen zum Primerdesign sind der Schlüssel zu einer spezifischen Amplifikation mit hoher Ausbeute. Die angegebenen Vorzugswerte sind in allen unseren Produkten standardmäßig eingebaut.
1. Primerlänge: Es ist allgemein anerkannt, dass die optimale Länge von PCR-Primern 18-22 bp beträgt. Diese Länge ist lang genug für eine ausreichende Spezifität und kurz genug, damit sich die Primer bei der Annealing-Temperatur leicht an die Vorlage binden können.
2. Primer-Schmelztemperatur: Die Primer-Schmelztemperatur (Tm) ist per Definition die Temperatur, bei der eine Hälfte des DNA-Duplexes dissoziiert und zu einem Einzelstrang wird, und gibt die Stabilität des Duplexes an. Primer mit Schmelztemperaturen im Bereich von 52-58 oC liefern im Allgemeinen die besten Ergebnisse. Primer mit Schmelztemperaturen über 65oC neigen zum sekundären Annealing. Der GC-Gehalt der Sequenz gibt einen guten Hinweis auf die Primer-Tm. Alle unsere Produkte berechnen die Tm nach der thermodynamischen Theorie der nächsten Nachbarn, die als weitaus bessere Methode zur Schätzung der Tm anerkannt ist und als die neueste und beste verfügbare Methode gilt.
Formel zur Berechnung der Primer-Tm:
Schmelztemperatur Tm(K)={ΔH/ ΔS + R ln(C)}, oder Schmelztemperatur Tm(oC) = {ΔH/ ΔS + R ln(C)} – 273.15 wobei
ΔH (kcal/mol) : H ist die Enthalpie. Die Enthalpie ist die Menge an Wärmeenergie, die ein Stoff besitzt. ΔH ist die Änderung der Enthalpie. In der obigen Formel erhält man ΔH, indem man die Enthalpiewerte aller Di-Nukleotidpaare der jeweils nächstgelegenen Basenpaare addiert.
ΔS (kcal/mol) : S ist der Grad der Unordnung, den ein System aufweist und wird Entropie genannt. ΔS ist die Veränderung der Entropie. In diesem Fall wird sie durch Addition aller Entropiewerte der Di-Nukleotidpaare der jeweils nächstgelegenen Basenpaare ermittelt. Eine zusätzliche Salzkorrektur wird hinzugefügt, da die Nearest Neighbor-Parameter aus DNA-Schmelzstudien stammen, die in 1M Na+-Puffer durchgeführt wurden, und dies die Standardbedingung für alle Berechnungen ist.
ΔS (Salzkorrektur) = ΔS (1M NaCl )+ 0.368 x N x ln()
Wobei
N die Anzahl der Nukleotidpaare im Primer ist ( Primerlänge -1).
ist Salzäquivalent in mM.
Berechnung:
= einwertige Ionenkonzentration +4 x freies Mg2+.
3. Primer-Schmelztemperatur: Die Schmelztemperatur des Primers ist ein Maß für die Stabilität des DNA-DNA-Hybrids und entscheidend für die Bestimmung der Annealing-Temperatur. Eine zu hohe Ta führt zu einer unzureichenden Primer-Template-Hybridisierung und damit zu einer geringen Ausbeute an PCR-Produkten. Eine zu niedrige Ta kann möglicherweise zu unspezifischen Produkten führen, die durch eine hohe Anzahl von Basenpaar-Fehlpaarungen verursacht werden. Die Mismatch-Toleranz hat den stärksten Einfluss auf die PCR-Spezifität.
Ta = 0,3 x Tm (Primer) + 0,7 Tm (Produkt) – 14.9
wobei,
Tm(Primer) = Schmelztemperatur der Primer
Tm(Produkt) = Schmelztemperatur des Produkts
4. GC-Gehalt: Der GC-Gehalt (die Anzahl der Gs und Cs im Primer als Prozentsatz der Gesamtbasen) des Primers sollte 40-60% betragen.
5. GC-Klammer: Das Vorhandensein von G- oder C-Basen innerhalb der letzten fünf Basen des 3′-Endes von Primern (GC-Clamp) fördert die spezifische Bindung am 3′-Ende aufgrund der stärkeren Bindung von G- und C-Basen. Mehr als 3 Gs oder Cs sollten in den letzten 5 Basen am 3′-Ende des Primers vermieden werden.
6. Primer-Sekundärstrukturen: Das Vorhandensein von Primer-Sekundärstrukturen, die durch inter- oder intramolekulare Wechselwirkungen entstehen, kann zu einer schlechten oder gar keiner Ausbeute des Produkts führen. Sie beeinträchtigen das Primer-Template-Annealing und damit die Amplifikation. Sie reduzieren die Verfügbarkeit der Primer für die Reaktion erheblich.
i) Haarnadeln: Sie werden durch intramolekulare Wechselwirkungen innerhalb des Primers gebildet und sollten vermieden werden. Optimal ist eine 3′-Endhaarnadel mit einem ΔG von -2 kcal/mol und eine Innenhaarnadel mit einem ΔG von -3 kcal/mol, die im Allgemeinen toleriert wird.
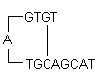
ΔG Definition: Die freie Gibbs-Energie G ist das Maß für die Arbeit, die einem Prozess bei konstantem Druck entzogen werden kann. Sie ist das Maß für die Spontaneität der Reaktion. Die Stabilität einer Haarnadel wird üblicherweise durch ihren ΔG-Wert angegeben, d. h. die Energie, die zum Aufbrechen der Sekundärstruktur erforderlich ist. Ein größerer negativer Wert für ΔG weist auf stabile, unerwünschte Haarnadeln hin. Das Vorhandensein von Haarnadeln am 3′-Ende beeinträchtigt die Reaktion am meisten.
ΔG = ΔH – TΔS
ii) Selbstdimer: Ein Primer-Selbstdimer wird durch intermolekulare Wechselwirkungen zwischen den beiden (gleichsinnigen) Primern gebildet, wobei der Primer zu sich selbst homolog ist. Im Allgemeinen wird bei der PCR eine große Menge an Primern im Vergleich zur Menge des Zielgens verwendet. Wenn Primer intermolekulare Dimere bilden, die viel leichter sind als die Hybridisierung mit der Ziel-DNA, verringern sie die Produktausbeute. Optimal ist ein 3′-End-Selbstdimer mit einem ΔG von -5 kcal/mol und ein internes Selbstdimer mit einem ΔG von -6 kcal/mol, das im Allgemeinen toleriert wird.
iii) Kreuzdimer: Primer-Kreuzdimere werden durch intermolekulare Wechselwirkung zwischen Sense- und Antisense-Primern gebildet, wenn sie homolog sind. Optimal ist ein Kreuzdimer am 3′-Ende mit einem ΔG von -5 kcal/mol und ein internes Kreuzdimer mit einem ΔG von -6 kcal/mol, das im Allgemeinen toleriert wird.
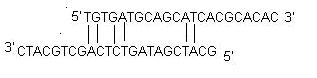
7. Wiederholungen: Ein Repeat ist ein Di-Nukleotid, das viele Male hintereinander vorkommt und vermieden werden sollte, da es zu Fehleinschätzungen kommen kann. Zum Beispiel: ATATATATAT. Die maximale Anzahl von Di-Nukleotid-Wiederholungen in einem Oligo beträgt 4 Di-Nukleotide.
8. Läufe: Primer mit langen Läufen einer einzelnen Base sollten generell vermieden werden, da sie misprime sein können. Zum Beispiel hat AGCGGGGGGATGGGG Läufe der Base ‚G‘ mit den Werten 5 und 4. Die maximale Anzahl der akzeptierten Läufe beträgt 4bp.
9. 3′ Endstabilität: Dies ist der maximale ΔG-Wert der fünf Basen des 3′-Endes. Ein instabiles 3′-Ende (weniger negatives ΔG) führt zu weniger falschem Priming.
10. Vermeiden Sie die Sekundärstruktur der Vorlage: Einzelsträngige Nukleinsäuresequenzen sind sehr instabil und falten sich zu Konformationen (Sekundärstrukturen). Die Stabilität dieser Template-Sekundärstrukturen hängt weitgehend von ihrer freien Energie und Schmelztemperatur (Tm) ab. Die Berücksichtigung der Sekundärstrukturen der Matrize ist beim Entwurf von Primern wichtig, insbesondere bei der qPCR. Werden Primer auf einer Sekundärstruktur entworfen, die selbst oberhalb der Annealing-Temperaturen stabil ist, können die Primer nicht an die Matrize binden, und die Ausbeute an PCR-Produkten wird erheblich beeinträchtigt. Daher ist es wichtig, Primer in den Bereichen der Vorlagen zu entwerfen, die während der PCR-Reaktion keine stabilen Sekundärstrukturen bilden. Unsere Produkte bestimmen die Sekundärstrukturen des Templates und entwerfen Primer, die diese vermeiden.
11. Vermeiden Sie Kreuzhomologie: Um die Spezifität der Primer zu verbessern, ist es notwendig, Regionen der Homologie zu vermeiden. Primer, die für eine Sequenz entwickelt wurden, dürfen keine anderen Gene in der Mischung amplifizieren. Üblicherweise werden Primer entworfen und dann mit BLAST überprüft, um die Spezifität zu testen. Unsere Produkte bieten eine bessere Alternative. Sie können beim Entwurf von Primern Regionen mit Kreuzhomologie vermeiden. Sie können die Vorlagen mit der entsprechenden nicht-redundanten Datenbank abgleichen (BLAST) und die Software interpretiert die Ergebnisse. Sie identifiziert Regionen mit signifikanter Kreuzhomologie in jeder Vorlage und vermeidet sie bei der Primersuche.
Parameter für das Primerpaar-Design
1. Amplikonlänge: Die Amplikonlänge wird von den Versuchszielen diktiert. Bei der qPCR liegt die Ziellänge bei etwa 100 bp, bei der Standard-PCR bei etwa 500 bp. Wenn Sie die Positionen der einzelnen Primer in Bezug auf die Vorlage kennen, wird das Produkt wie folgt berechnet: Produktlänge = (Position des Antisense-Primers – Position des Sense-Primers) + 1.
2. Produktposition: Der Primer kann sich in der Nähe des 5′-Endes, des 3′-Endes oder an einer beliebigen Stelle innerhalb der angegebenen Länge befinden. Im Allgemeinen ist die Sequenz nahe dem 3′-Ende mit größerer Sicherheit bekannt und wird daher am häufigsten bevorzugt.
3. Tm des Produkts: Die Schmelztemperatur (Tm) ist die Temperatur, bei der eine Hälfte des DNA-Duplexes dissoziiert und einzelsträngig wird. Die Stabilität des Primer-Template-DNA-Duplex kann anhand der Schmelztemperatur (Tm) gemessen werden.
4. Optimale Annealing-Temperatur (Ta Opt): Die Formel von Rychlik ist die am meisten beachtete. Unsere Produkte verwenden diese Formel, um sie zu berechnen, und Tausende unserer Kunden haben über gute Ergebnisse berichtet, wenn sie sie für den Annealing-Schritt des PCR-Zyklus verwenden. Sie führt in der Regel zu einer guten PCR-Produktausbeute mit minimaler Fehlproduktproduktion.
Ta Opt = 0.3 x(Tm des Primers) + 0.7 x(Tm des Produkts) – 14.9
wobei
Tm des Primers die Schmelztemperatur des weniger stabilen Primer-Template-Paares
Tm des Produktes die Schmelztemperatur des PCR-Produktes ist.
5. Primer Pair Tm Mismatch Calculation: Die beiden Primer eines Primerpaares sollten eng aufeinander abgestimmte Schmelztemperaturen haben, um die Ausbeute an PCR-Produkten zu maximieren. Ein Unterschied von 5oC oder mehr kann zu keiner Amplifikation führen.
Primerdesign mit Software
Es gibt eine Reihe von Primerdesign-Tools, die sowohl neuen als auch erfahrenen Anwendern beim PCR-Primerdesign helfen können. Diese Werkzeuge können die Kosten und den Zeitaufwand für Experimente reduzieren, indem sie die Wahrscheinlichkeit von Fehlversuchen verringern.
Primer Premier folgt allen Richtlinien, die für das Design von PCR-Primern festgelegt wurden. Primer Premier kann für das Design von Primern für einzelne Templates, Alignments, das Design degenerierter Primer, Restriktionsenzymanalysen, Contig-Analysen und das Design von Sequenzierungsprimern verwendet werden.
Die Richtlinien für das Design von qPCR-Primern variieren leicht. Software wie AlleleID und Beacon Designer können Primer und Oligonukleotid-Sonden für komplexe Nachweisverfahren wie Multiplex-Assays, artübergreifendes Primerdesign, artspezifisches Primerdesign und Primerdesign zur Senkung der Experimentierkosten entwerfen.
PrimerPlex ist eine Software, mit der Primer für Multiplex-PCR- und Multiplex-SNP-Genotypisierungsassays entworfen werden können.