Referințe >
PCR (Reacția în lanț a polimerazei)
Reacția în lanț a polimerazei este considerată pe scară largă ca fiind una dintre cele mai importante invenții ale secolului XX în domeniul biologiei moleculare. Cantități mici de material genetic pot fi acum amplificate pentru a putea a identifica, manipula ADN-ul, detecta organisme infecțioase, inclusiv virușii care provoacă SIDA, hepatita, tuberculoza, detecta variații genetice, inclusiv mutații, în genele umane și numeroase alte sarcini.
PCR implică următoarele trei etape: Denaturarea, recoacerea și extensia. În primul rând, materialul genetic este denaturat, transformând moleculele de ADN bicatenar în monocatenar. Amorsele sunt apoi aneantizate la regiunile complementare ale moleculelor monocatenare. În a treia etapă, acestea sunt extinse prin acțiunea ADN polimerazei. Toate aceste etape sunt sensibile la temperatură, iar alegerea obișnuită a temperaturilor este de 94oC, 60oC și, respectiv, 70oC. O bună proiectare a amorselor este esențială pentru succesul reacțiilor. Considerentele importante de proiectare descrise mai jos reprezintă o cheie pentru o amplificare specifică cu randament ridicat. Valorile preferate indicate sunt încorporate implicit în toate produsele noastre.
1. Lungimea amorselor: Este în general acceptat faptul că lungimea optimă a primerilor PCR este de 18-22 bp. Această lungime este suficient de lungă pentru o specificitate adecvată și suficient de scurtă pentru ca amorsele să se lege ușor de șablon la temperatura de aneantizare.
2. Temperatura de topire a amorsei: Temperatura de topire a amorselor (Tm) prin definiție este temperatura la care o jumătate din duplexul de ADN se va disocia pentru a deveni monocatenar și indică stabilitatea duplexului. Amorsele cu temperaturi de topire în intervalul 52-58 oC produc, în general, cele mai bune rezultate. Amorsele cu temperaturi de topire de peste 65 oC au tendința de aneantizare secundară. Conținutul de GC al secvenței oferă o indicație corectă a Tm a amorselor. Toate produsele noastre o calculează folosind teoria termodinamică a vecinului cel mai apropiat, acceptată ca o metodă mult superioară de estimare a acesteia, care este considerată cea mai recentă și cea mai bună metodă disponibilă.
Formula pentru calcularea Tm a amorselor:
Temperatura de topire Tm(K)={ΔH/ ΔS + R ln(C)}, Sau temperatura de topire Tm(oC) = {ΔH/ ΔS + R ln(C)} – 273,15 unde
ΔH (kcal/mole) : H este Entalpia. Entalpia este cantitatea de energie termică pe care o posedă substanțele. ΔH este variația Entalpiei. În formula de mai sus, ΔH se obține prin însumarea tuturor valorilor entalpiei perechilor de di-nucleotide ale fiecărei perechi de baze cele mai apropiate.
ΔS (kcal/mole) : S este cantitatea de dezordine pe care o prezintă un sistem se numește entropie. ΔS este modificarea entropiei. Aici se obține prin însumarea tuturor valorilor entropiei perechilor de di-nucleotide ale fiecărei perechi de baze cel mai apropiat vecin. Se adaugă o corecție suplimentară pentru sare, deoarece parametrii de vecinătate cea mai apropiată au fost obținuți din studii de topire a ADN-ului efectuate în soluție tampon 1M Na+ și aceasta este condiția implicită utilizată pentru toate calculele.
ΔS (corecție pentru sare) = ΔS (1M NaCl )+ 0.368 x N x ln()
Unde
N este numărul de perechi de nucleotide din amorsă ( lungime amorsă -1).
este echivalentul de sare în mM.
calcul:
= Concentrația de ioni monovalenți +4 x Mg2+ liber.
3. Temperatura de recoacere a amorselor: Temperatura de topire a amorselor este o estimare a stabilității hibridului ADN-ADN și este esențială pentru determinarea temperaturii de recoacere. O Ta prea mare va produce o hibridizare insuficientă amorsă-șablon, rezultând un randament scăzut al produsului PCR. O Ta prea scăzută poate duce, eventual, la produse nespecifice cauzate de un număr mare de nepotriviri de perechi de baze,. S-a constatat că toleranța la nepotriviri are cea mai mare influență asupra specificității PCR.
Ta = 0,3 x Tm(primer) + 0,7 Tm (produs) – 14.9
unde,
Tm(primer) = Temperatura de topire a primerilor
Tm(produs) = Temperatura de topire a produsului
4. Conținutul GC: Conținutul de GC (numărul de G-uri și C-uri din amorsă ca procent din totalul bazelor) al amorselor trebuie să fie de 40-60%.
5. Clema GC: Prezența bazelor G sau C în ultimele cinci baze de la capătul 3′ al amorselor (GC clamp) ajută la promovarea legăturii specifice la capătul 3′ datorită legăturii mai puternice a bazelor G și C. Trebuie evitate mai mult de 3 G sau C în ultimele 5 baze de la capătul 3′ al amorselor.
6. Structuri secundare ale amorselor: Prezența structurilor secundare ale amorselor produse de interacțiunile intermoleculare sau intramoleculare poate duce la un randament slab sau la absența produsului. Acestea afectează în mod negativ aneantizarea șablonului de amorsă și, prin urmare, amplificarea. Acestea reduc foarte mult disponibilitatea amorselor pentru reacție.
i) Hairpins: Se formează prin interacțiune intramoleculară în cadrul amorselor și trebuie evitate. În mod optim se tolerează în general un hairpin la capătul 3′ cu un ΔG de -2 kcal/mol și un hairpin intern cu un ΔG de -3 kcal/mol.
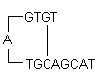
Definirea ΔG: Energia liberă Gibbs G este măsura cantității de lucru care poate fi extrasă dintr-un proces care funcționează la o presiune constantă. Este măsura spontaneității reacției. Stabilitatea unui ac de păr este reprezentată în mod obișnuit prin valoarea ΔG, energia necesară pentru a rupe structura secundară. O valoare negativă mai mare pentru ΔG indică o hairpin stabilă, nedorită. Prezența de hairpins la capătul 3′ afectează cel mai negativ reacția.
ΔG = ΔH – TΔS
ii) Autodimerul: Un autodimer de amorsă se formează prin interacțiuni intermoleculare între cei doi amors (cu același sens), în cazul în care amorsa este omologă cu ea însăși. În general, în PCR se utilizează o cantitate mare de amorse în comparație cu cantitatea de genă țintă. Atunci când amorsele formează dimeri intermoleculari mult mai ușor decât să se hibrideze cu ADN-ul țintă, acestea reduc randamentul produsului. În mod optim se tolerează, în general, un autodimer la capătul 3′ cu un ΔG de -5 kcal/mol și un autodimer intern cu un ΔG de -6 kcal/mol.
iii) Dimer încrucișat: Dimerii încrucișați ai amorselor se formează prin interacțiunea intermoleculară între amorsă sens și antisens, atunci când acestea sunt omoloage. În mod optim, se tolerează în general un dimer încrucișat la capătul 3′ cu un ΔG de -5 kcal/mol și un dimer încrucișat intern cu un ΔG de -6 kcal/mol.
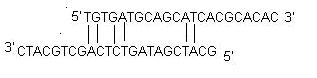
7. Repetiții: O repetiție este o di-nucleotidă care apare de mai multe ori consecutiv și ar trebui evitată, deoarece acestea pot să primeze greșit. De exemplu: ATATATATAT. Un număr maxim de repetări de di-nucleotide acceptabile într-un oligo este de 4 di-nucleotide.
8. Runs: În general, trebuie evitați amperele cu runde lungi de o singură bază, deoarece acestea se pot suprapune greșit. De exemplu, AGCGGGGGGGATGGGGGG are curse de bază „G” de valoare 5 și 4. Un număr maxim de run-uri acceptate este de 4bp.
9. Stabilitatea capătului 3′: Este valoarea ΔG maximă a celor cinci baze de la capătul 3′. O extremitate 3′ instabilă (ΔG mai puțin negativă) va duce la mai puțină amorsare falsă.
10. Evitați structura secundară a șablonului: O secvență monocatenară de acid nucleic este foarte instabilă și se pliază în conformații (structuri secundare). Stabilitatea acestor structuri secundare de șablon depinde în mare măsură de energia lor liberă și de temperaturile de topire(Tm). Luarea în considerare a structurilor secundare ale șablonului este importantă în proiectarea amorselor, în special în qPCR. În cazul în care amorsele sunt proiectate pe o structură secundară care este stabilă chiar și peste temperaturile de aneantizare, amorsele nu se pot lega de șablon, iar randamentul produsului PCR este afectat în mod semnificativ. Prin urmare, este important să se proiecteze primeri în regiunile din șabloane care nu formează structuri secundare stabile în timpul reacției PCR. Produsele noastre determină structurile secundare ale șablonului și proiectează amorse care le evită.
11. Evitați omologia încrucișată: Pentru a îmbunătăți specificitatea amorselor este necesar să se evite regiunile de omologie. Amorsele concepute pentru o secvență nu trebuie să amplifice alte gene din amestec. În mod obișnuit, se proiectează primeri și apoi se testează prin BLAST pentru a testa specificitatea. Produsele noastre oferă o alternativă mai bună. Puteți evita regiunile de omologie încrucișată în timpul proiectării amorselor. Puteți compara șabloanele prin BLAST cu baza de date neredundantă corespunzătoare, iar software-ul va interpreta rezultatele. Acesta va identifica regiunile de omologii încrucișate semnificative în fiecare șablon și le va evita în timpul căutării amorselor.
Parametri pentru proiectarea perechilor de amorsă
1. Lungimea ampliconului: Lungimea ampliconului este dictată de obiectivele experimentale. Pentru qPCR, lungimea țintă este mai aproape de 100 pb, iar pentru PCR standard, aceasta se apropie de 500 pb. Dacă cunoașteți pozițiile fiecărui amorsă în raport cu șablonul, produsul se calculează astfel: Lungimea produsului = (Poziția amorsă antisens-Poziția amorsă sens) + 1.
2. Poziția produsului: Primerul poate fi localizat lângă capătul 5′, capătul 3′ sau oriunde în cadrul lungimii specificate. În general, secvența apropiată de capătul 3′ este cunoscută cu mai multă încredere și, prin urmare, este preferată cel mai frecvent.
3. Tm a produsului: Temperatura de topire (Tm) este temperatura la care o jumătate din duplexul de ADN se va disocia și va deveni monocatenar. Stabilitatea duplexului amorsă-șablon de ADN poate fi măsurată prin temperatura de topire (Tm).
4. Temperatura optimă de recoacere (Ta Opt): Formula lui Rychlik este cea mai respectată. Produsele noastre folosesc această formulă pentru a o calcula și mii de clienți au raportat rezultate bune folosind-o pentru etapa de recoacere a ciclului PCR. De obicei, aceasta are ca rezultat un randament bun al produsului PCR cu o producție minimă de produse false.
Ta Opt = 0,3 x(Tm a primerului) + 0,7 x(Tm a produsului) – 14.9
unde
Tm de amorsă este temperatura de topire a perechii amorsă-șablon mai puțin stabile
Tm de produs este temperatura de topire a produsului PCR.
5. Calcularea nepotrivirii Tm a perechii de amorsă: Cei doi primeri ai unei perechi de primeri trebuie să aibă temperaturi de topire foarte apropiate pentru a maximiza randamentul produsului PCR. Diferența de 5oC sau mai mult poate duce la lipsa amplificării.
Proiectarea amorselor cu ajutorul unui software
Există o serie de instrumente de proiectare a amorselor care pot ajuta la proiectarea amorselor PCR atât pentru utilizatorii noi, cât și pentru cei experimentați. Aceste instrumente pot reduce costurile și timpul implicat în experimentare, scăzând șansele de eșec al experimentelor.
Primer Premier respectă toate liniile directoare specificate pentru proiectarea primerilor PCR. Primer Premier poate fi utilizat pentru a proiecta amorse pentru șabloane unice, alinieri, proiectarea de amorse degenerate, analiza enzimelor de restricție. analiza contigurilor și proiectarea de amorse de secvențiere.
Legitimările pentru proiectarea de amorse qPCR variază ușor. Softuri precum AlleleID și Beacon Designer pot proiecta amorse și sonde oligonucleotidice pentru teste de detecție complexe, cum ar fi testele multiplex, proiectarea de amorse pentru specii încrucișate, proiectarea de amorse specifice speciilor și proiectarea de amorse pentru a reduce costurile de experimentare.
PrimerPlex este un software care poate proiecta amorse pentru testele de PCR multiplex și de genotipare SNP multiplex.
.