B. Replicación del ADN en las bacterias
En general, el ADN se replica por desenrollamiento de la hélice, separación de la hebra por ruptura de los enlaces de hidrógeno entre las hebras complementarias, y síntesis de dos nuevas hebras por emparejamiento de bases complementarias. La replicación comienza en un sitio específico del ADN llamado origen de replicación (oriC).
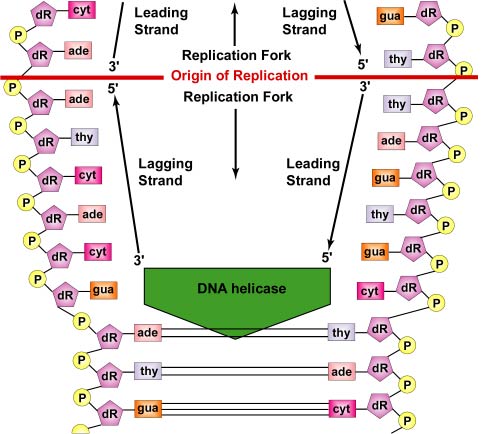
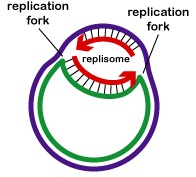
La replicación del ADN es bidireccional desde el origen de replicación. Para comenzar la replicación del ADN, unas enzimas desenrolladoras llamadas helicasas de ADN hacen que segmentos cortos de las dos hebras de ADN padre se desenrollen y se separen entre sí en el origen de la replicación para formar dos horquillas de replicación en forma de «Y». Estas horquillas de replicación son el lugar real de la copia del ADN (Figura \(\PageIndex{3}\)). Todas las proteínas implicadas en la replicación del ADN se agrupan en las horquillas de replicación para formar un complejo de replicación llamado replisoma (Figura \(\PageIndex{4}\)).
Las proteínas de unión de la cadena simple se unen a las regiones de la cadena simple para que las dos cadenas no se vuelvan a unir. El desenrollado de la hélice de doble cadena genera superenrollamientos positivos por delante de la horquilla de replicación. Unas enzimas denominadas topoisomerasas contrarrestan esta situación produciendo roturas en el ADN y volviéndolas a unir para formar superenrollamientos negativos con el fin de aliviar esta tensión en la molécula helicoidal durante la replicación.
A medida que las hebras continúan desenrollándose y separándose en ambas direcciones alrededor de toda la molécula de ADN, se producen nuevas hebras complementarias mediante el enlace de hidrógeno de los nucleótidos libres del ADN con los de cada hebra madre. A medida que los nuevos nucleótidos se alinean frente a cada hebra madre mediante enlaces de hidrógeno, las enzimas llamadas ADN polimerasas unen los nucleótidos mediante enlaces fosfodiéster. En realidad, los nucleótidos que se alinean por emparejamiento de bases complementarias son trifosfatos de desoxinucleótidos, compuestos por una base nitrogenada, desoxirribosa y tres fosfatos. A medida que se forma el enlace fosfodiéster entre el grupo fosfato 5′ del nuevo nucleótido y el OH 3′ del último nucleótido de la cadena de ADN, dos de los fosfatos se eliminan proporcionando energía para el enlace (véase la Figura \(\PageIndex{6})). Al final, cada hebra madre sirve como plantilla para sintetizar una copia complementaria de sí misma, dando lugar a la formación de dos moléculas de ADN idénticas (ver Figura \(\PageIndex{7}\)). En las bacterias, las proteínas Par funcionan para separar los cromosomas bacterianos en polos opuestos de la célula durante la división celular. Se unen al origen de replicación del ADN y separan físicamente los cromosomas, de forma similar al aparato mitótico de las células eucariotas. Las proteínas Fts, como la FtsK en el divisoma, también ayudan a separar el cromosoma bacteriano replicado.
Animación GIF que ilustra la replicación del ADN por emparejamiento de bases complementarias
En realidad, la replicación del ADN es más complicada que esto debido a la naturaleza de las ADN polimerasas. Las enzimas ADN polimerasas sólo son capaces de unir el grupo fosfato del carbono 5′ de un nuevo nucleótido con el grupo hidroxilo (OH) del carbono 3′ de un nucleótido ya existente en la cadena. Como resultado, el ADN sólo puede sintetizarse en dirección 5′ a 3′ mientras se copia una cadena madre que va en dirección 3′ a 5′.
Cada cadena de ADN tiene dos extremos. El extremo 5′ del ADN es el que tiene el grupo fosfato terminal en el carbono 5′ de la desoxirribosa; el extremo 3′ es el que tiene un grupo hidroxilo (OH) terminal en la desoxirribosa del carbono 3′ de la desoxirribosa (ver Figura \(\PageIndex{8}\)). Las dos hebras son antiparalelas, es decir, van en direcciones opuestas. Por lo tanto, una de las cadenas madre – la que va de 3′ a 5′ y que se denomina cadena principal – puede copiarse directamente a lo largo de toda su longitud (véase la figura (índice de página 9)). Sin embargo, la otra hebra madre -la que va de 5′ a 3′ y que se denomina hebra rezagada- debe copiarse de forma discontinua en fragmentos cortos (fragmentos de Okazaki) de unos 100-1000 nucleótidos cada uno a medida que el ADN se desenrolla. Esto ocurre, como se ha mencionado anteriormente, en el replisoma. La hebra de ADN retrasada se separa de la hebra principal y esto permite que el replisoma se mueva a lo largo de ambas hebras tirando del ADN a medida que se produce la replicación. Es el propio ADN, y no la ADN polimerasa, el que se mueve durante la replicación del ADN bacteriano (ver Figura \(\PageIndex{5})).
Además, las enzimas de la ADN polimerasa no pueden comenzar una nueva cadena de ADN desde cero. Sólo pueden unir nuevos nucleótidos al grupo 3′ OH de un nucleótido en una cadena preexistente. Por lo tanto, para iniciar la síntesis de la cadena principal y de cada fragmento de ADN de la cadena rezagada, se requiere un complejo de ARN polimerasa llamado primasa. La primasa, que es capaz de unir nucleótidos de ARN sin requerir una hebra preexistente de ácido nucleico, añade primero varios nucleótidos de ARN complementarios frente a los nucleótidos de ADN de la hebra madre. Esto forma lo que se llama un cebador de ARN (ver Figura \(\PageIndex{10}\)).
La ADN polimerasa III sustituye entonces a la primasa y es capaz de añadir nucleótidos de ADN al cebador de ARN (ver Figura \(\PageIndex{11}\)). Posteriormente, la ADN polimerasa II digiere el cebador de ARN y sustituye los nucleótidos de ARN del cebador por los nucleótidos de ADN adecuados para rellenar el hueco (ver Figura \(\PageIndex{12}\)). Por último, los fragmentos de ADN se unen mediante la enzima ADN ligasa (véase la Figura \(\PageIndex{9}\)). Sin embargo, incluso con este complicado procedimiento, una macromolécula de 1000 micrómetros de largo de ADN fuertemente empaquetado y superenrollado puede hacer una copia exacta de sí mismo en sólo unos 10 minutos de tiempo en condiciones óptimas, la inserción de nucleótidos a una velocidad de alrededor de 1000 nucleótidos por segundo!
Animación de la replicación del ADN.
Cortesía de Biointeractive del HHMI.