Referencias >> Guía de diseño de cebadores para la PCR
PCR (reacción en cadena de la polimerasa)
La reacción en cadena de la polimerasa es ampliamente considerada como uno de los inventos más importantes del siglo XX en biología molecular. Ahora se pueden amplificar pequeñas cantidades de material genético para poder identificar, manipular el ADN, detectar organismos infecciosos, incluyendo los virus que causan el SIDA, la hepatitis, la tuberculosis, detectar variaciones genéticas, incluyendo mutaciones, en los genes humanos y otras numerosas tareas.
La PCR implica los siguientes tres pasos: Desnaturalización, Recalentamiento y Extensión. En primer lugar, se desnaturaliza el material genético, convirtiendo las moléculas de ADN de doble cadena en cadenas simples. A continuación, los cebadores se recobran en las regiones complementarias de las moléculas monocatenarias. En el tercer paso, se extienden por la acción de la ADN polimerasa. Todos estos pasos son sensibles a la temperatura y la elección común de las temperaturas es 94oC, 60oC y 70oC respectivamente. Un buen diseño de los cebadores es esencial para el éxito de las reacciones. Las importantes consideraciones de diseño que se describen a continuación son la clave para una amplificación específica con un alto rendimiento. Los valores preferidos indicados están incorporados por defecto en todos nuestros productos.
1. Longitud del cebador: En general se acepta que la longitud óptima de los cebadores de PCR es de 18-22 pb. Esta longitud es lo suficientemente larga para una especificidad adecuada y lo suficientemente corta para que los cebadores se unan fácilmente al molde a la temperatura de recocido.
2. Temperatura de fusión del cebador: La temperatura de fusión de los cebadores (Tm) es, por definición, la temperatura a la que una mitad del dúplex de ADN se disociará para convertirse en una sola hebra e indica la estabilidad del dúplex. Los cebadores con temperaturas de fusión en el rango de 52-58 oC generalmente producen los mejores resultados. Los cebadores con temperaturas de fusión superiores a 65oC tienen tendencia a la recocido secundario. El contenido de GC de la secuencia da una buena indicación de la Tm del cebador. Todos nuestros productos la calculan utilizando la teoría termodinámica del vecino más cercano, aceptada como un método muy superior para estimarla, que se considera el más reciente y el mejor disponible.
Fórmula para el cálculo de la Tm del cebador:
Temperatura de fusión Tm(K)={ΔH/ ΔS + R ln(C)}, O Temperatura de fusión Tm(oC) = {ΔH/ ΔS + R ln(C)} – 273.15 donde
ΔH (kcal/mole) : H es la Entalpía. La entalpía es la cantidad de energía calorífica que poseen las sustancias. ΔH es el cambio en la entalpía. En la fórmula anterior la ΔH se obtiene sumando todos los valores de entalpía de los pares de di-nucleótidos de cada par de bases vecino más cercano.
ΔS (kcal/mole) : S es la cantidad de desorden que presenta un sistema se llama entropía. ΔS es el cambio en la Entropía. Aquí se obtiene sumando todos los valores de entropía de los pares de di-nucleótidos de cada par de bases vecino más cercano. Se añade una corrección de sal adicional ya que los parámetros de Vecino más cercano se obtuvieron a partir de estudios de fusión de ADN realizados en tampón 1M Na+ y esta es la condición por defecto utilizada para todos los cálculos.
ΔS (corrección de sal) = ΔS (1M NaCl )+ 0.368 x N x ln()
Donde
N es el número de pares de nucleótidos en el cebador ( longitud del cebador -1).
es el equivalente salino en mM.
cálculo:
= concentración de iones monovalentes +4 x Mg2+ libre.
3. Temperatura de recocido del primer: La temperatura de fusión del primer es la estimación de la estabilidad del híbrido ADN-ADN y es crítica para determinar la temperatura de recocido. Una Ta demasiado alta producirá una hibridación insuficiente entre el cebador y la placa, lo que dará lugar a un bajo rendimiento del producto de la PCR. Una Ta demasiado baja puede dar lugar a productos no específicos causados por un elevado número de desajustes de pares de bases. La tolerancia a los desajustes es la que más influye en la especificidad de la PCR.
Ta = 0,3 x Tm(cebador) + 0,7 Tm (producto) – 14.9
donde,
Tm(primer) = Temperatura de fusión de los primers
Tm(producto) = Temperatura de fusión del producto
4. Contenido de GC: El contenido de GC (el número de G’s y C’s en el cebador como porcentaje de las bases totales) del cebador debe ser del 40-60%.
5. Pinza de GC: La presencia de bases G o C dentro de las últimas cinco bases del extremo 3′ de los cebadores (GC clamp) ayuda a promover la unión específica en el extremo 3′ debido a la unión más fuerte de las bases G y C. Deben evitarse más de 3 G o C en las últimas 5 bases del extremo 3′ del cebador.
6. Estructuras secundarias del cebador: La presencia de las estructuras secundarias del cebador producidas por interacciones intermoleculares o intramoleculares puede conducir a un rendimiento pobre o nulo del producto. Afectan negativamente al recocido de la plantilla del cebador y, por tanto, a la amplificación. Reducen en gran medida la disponibilidad de los cebadores para la reacción.
i) Horquillas: Se forman por interacción intramolecular dentro del cebador y deben evitarse. De forma óptima se tolera generalmente una horquilla en el extremo 3′ con un ΔG de -2 kcal/mol y una horquilla interna con un ΔG de -3 kcal/mol.
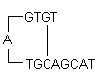
ΔG definición: La energía libre de Gibbs G es la medida de la cantidad de trabajo que se puede extraer de un proceso que opera a una presión constante. Es la medida de la espontaneidad de la reacción. La estabilidad de la horquilla se suele representar por su valor de ΔG, la energía necesaria para romper la estructura secundaria. Un valor negativo mayor de ΔG indica la existencia de horquillas estables e indeseables. La presencia de horquillas en el extremo 3′ afecta más negativamente a la reacción.
ΔG = ΔH – TΔS
ii) Autodímero: Un autodímero de cebador se forma por interacciones intermoleculares entre los dos cebadores (del mismo sentido), donde el cebador es homólogo a sí mismo. Por lo general, en la PCR se utiliza una gran cantidad de cebadores en comparación con la cantidad del gen objetivo. Cuando los cebadores forman dímeros intermoleculares con mayor facilidad que la hibridación con el ADN diana, reducen el rendimiento del producto. De manera óptima se tolera generalmente un dímero propio del extremo 3′ con una ΔG de -5 kcal/mol y un dímero propio interno con una ΔG de -6 kcal/mol.
iii) Dímero cruzado: Los dímeros cruzados de los cebadores se forman por la interacción intermolecular entre los cebadores sentido y antisentido, cuando son homólogos. De forma óptima se tolera generalmente un dímero cruzado en el extremo 3′ con una ΔG de -5 kcal/mol y un dímero cruzado interno con una ΔG de -6 kcal/mol.
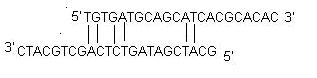
7. Repeticiones: Una repetición es un di-nucleótido que se produce muchas veces consecutivas y debe evitarse porque pueden dar lugar a errores de impronta. Por ejemplo: ATATATAT. El número máximo de repeticiones de di-nucleótidos aceptable en un oligo es de 4 di-nucleótidos.
8. Recorridos: Los cebadores con recorridos largos de una sola base deben evitarse en general, ya que pueden dar lugar a errores de impronta. Por ejemplo, AGCGGGGATGGG tiene corridas de base ‘G’ de valor 5 y 4. El número máximo de carreras aceptado es de 4bp.
9. Estabilidad del extremo 3′: Es el valor máximo de ΔG de las cinco bases del extremo 3′. Un extremo 3′ inestable (menos ΔG negativo) dará lugar a un menor cebado falso.
10. Evitar la estructura secundaria de la plantilla: Una secuencia de ácido nucleico monocatenario es altamente inestable y se pliega en conformaciones (estructuras secundarias). La estabilidad de estas estructuras secundarias de la plantilla depende en gran medida de su energía libre y de las temperaturas de fusión (Tm). La consideración de las estructuras secundarias del molde es importante en el diseño de los cebadores, especialmente en la qPCR. Si los cebadores se diseñan sobre estructuras secundarias que son estables incluso por encima de las temperaturas de recocido, los cebadores son incapaces de unirse al molde y el rendimiento del producto de la PCR se ve significativamente afectado. Por lo tanto, es importante diseñar los cebadores en las regiones de las plantillas que no forman estructuras secundarias estables durante la reacción de PCR. Nuestros productos determinan las estructuras secundarias de la plantilla y diseñan los cebadores evitándolas.
11. Evitar la homología cruzada: Para mejorar la especificidad de los cebadores es necesario evitar las regiones de homología. Los cebadores diseñados para una secuencia no deben amplificar otros genes de la mezcla. Comúnmente, los cebadores se diseñan y luego se hace un BLAST para probar la especificidad. Nuestros productos ofrecen una alternativa mejor. Puede evitar las regiones de homología cruzada al diseñar los cebadores. Puede realizar un BLAST de las plantillas contra la base de datos no redundante apropiada y el software interpretará los resultados. Identificará las regiones de homologías cruzadas significativas en cada plantilla y las evitará durante la búsqueda de primers.
Parámetros para el diseño de pares de primers
1. Longitud del amplicón: La longitud del amplicón viene dictada por los objetivos experimentales. Para la qPCR, la longitud objetivo es más cercana a 100 pb y para la PCR estándar, es cercana a 500 pb. Si se conocen las posiciones de cada cebador con respecto al molde, el producto se calcula como Longitud del producto = (Posición del primer antisentido-Posición del primer sentido) + 1.
2. Posición del producto: El cebador puede estar situado cerca del extremo 5′, del extremo 3′ o en cualquier lugar dentro de la longitud especificada. Generalmente, la secuencia cercana al extremo 3′ se conoce con mayor confianza y por ello se prefiere con mayor frecuencia.
3. Tm del Producto: La temperatura de fusión (Tm) es la temperatura a la que una mitad del dúplex de ADN se disociará y se convertirá en una sola hebra. La estabilidad del dúplex de ADN cebador-plantilla se puede medir por la temperatura de fusión (Tm).
4. Temperatura óptima de recocido (Ta Opt): La fórmula de Rychlik es la más respetada. Nuestros productos utilizan esta fórmula para calcularla y miles de nuestros clientes han reportado buenos resultados utilizándola para el paso de recocido del ciclo de PCR. Suele dar un buen rendimiento del producto de PCR con una mínima producción de falso producto.
Ta Opt = 0,3 x(Tm del cebador) + 0,7 x(Tm del producto) – 14.9
donde
Tm del cebador es la temperatura de fusión del par cebador-plantilla menos estable
Tm del producto es la temperatura de fusión del producto de la PCR.
5. Cálculo del desajuste de la Tm del par de cebadores: Los dos cebadores de un par de cebadores deben tener temperaturas de fusión muy parecidas para maximizar el rendimiento del producto de la PCR. La diferencia de 5oC o más puede hacer que no haya amplificación.
Diseño de cebadores mediante software
Existen varias herramientas de diseño de cebadores que pueden ayudar en el diseño de cebadores de PCR tanto a los usuarios nuevos como a los experimentados. Estas herramientas pueden reducir el coste y el tiempo que conlleva la experimentación, disminuyendo las posibilidades de experimentación fallida.
Primer Premier sigue todas las directrices especificadas para el diseño de cebadores PCR. Primer Premier puede utilizarse para diseñar cebadores para plantillas individuales, alineaciones, diseño de cebadores degenerados, análisis de enzimas de restricción. análisis contig y diseño de cebadores de secuenciación.
Las directrices para el diseño de cebadores de qPCR varían ligeramente. Los programas informáticos como AlleleID y Beacon Designer pueden diseñar cebadores y sondas de oligonucleótidos para ensayos de detección complejos como los ensayos multiplex, el diseño de cebadores de especies cruzadas, el diseño de cebadores específicos de especies y el diseño de cebadores para reducir el coste de la experimentación.
PrimerPlex es un programa informático que puede diseñar cebadores para la PCR multiplex y los ensayos de genotipado SNP multiplex.