B. Réplication de l’ADN chez les bactéries
En général, l’ADN se réplique par le déroulage de l’hélice, la séparation des brins par rupture des liaisons hydrogène entre les brins complémentaires, et la synthèse de deux nouveaux brins par appariement de bases complémentaires. La réplication commence à un site spécifique de l’ADN appelé origine de réplication (oriC).
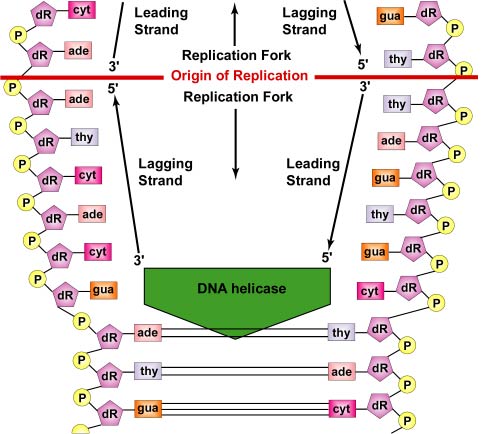
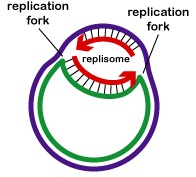
La réplication de l’ADN est bidirectionnelle à partir de l’origine de la réplication. Pour commencer la réplication de l’ADN, des enzymes de déroulement appelées ADN hélicases font en sorte que de courts segments des deux brins d’ADN parents se déroulent et se séparent l’un de l’autre à l’origine de la réplication pour former deux fourches de réplication en forme de « Y ». Ces fourches de réplication sont le site même de la copie de l’ADN (Figure \(\PageIndex{3}\)). Toutes les protéines impliquées dans la réplication de l’ADN s’agrègent au niveau des fourches de réplication pour former un complexe de réplication appelé réplisome (figure \(\PageIndex{4}\)).
Les protéines de liaison simple brin se fixent aux régions simple brin afin que les deux brins ne se rejoignent pas. Le déroulement de l’hélice double brin génère des super bobines positives en amont de la fourche de réplication. Des enzymes appelées topoisomérases contrecarrent ce phénomène en produisant des cassures dans l’ADN, puis les rejoignent pour former des super bobines négatives afin de soulager ce stress dans la molécule hélicoïdale pendant la réplication.
Alors que les brins continuent à se dérouler et à se séparer dans les deux sens autour de la molécule d’ADN entière, de nouveaux brins complémentaires sont produits par la liaison hydrogène des nucléotides libres de l’ADN avec ceux de chaque brin parent. Lorsque les nouveaux nucléotides s’alignent en face de chaque brin parent par liaison hydrogène, des enzymes appelées ADN polymérases relient les nucléotides par des liaisons phosphodiesters. En fait, les nucléotides qui s’alignent par appariement de bases complémentaires sont des désoxynucléotides triphosphates, composés d’une base azotée, le désoxyribose, et de trois phosphates. Lorsque la liaison phosphodiester se forme entre le groupe phosphate 5′ du nouveau nucléotide et le groupe OH 3′ du dernier nucléotide du brin d’ADN, deux des phosphates sont éliminés, fournissant ainsi l’énergie nécessaire à la liaison (voir figure \(\PageIndex{6}\)). Au final, chaque brin parent sert de matrice pour synthétiser une copie complémentaire de lui-même, ce qui aboutit à la formation de deux molécules d’ADN identiques (voir figure \(\PageIndex{7}\)). Chez les bactéries, les protéines Par ont pour fonction de séparer les chromosomes bactériens aux pôles opposés de la cellule pendant la division cellulaire. Elles se lient à l’origine de la réplication de l’ADN et séparent ou poussent physiquement les chromosomes, comme le fait l’appareil mitotique des cellules eucaryotes. Les protéines Fts, telles que FtsK dans le divisome, aident également à séparer le chromosome bactérien répliqué.
Une animation GIF illustrant la réplication de l’ADN par appariement de bases complémentaires
En réalité, la réplication de l’ADN est plus compliquée que cela en raison de la nature des ADN polymérases. Les enzymes ADN polymérases sont uniquement capables de joindre le groupe phosphate du carbone 5′ d’un nouveau nucléotide au groupe hydroxyle (OH) du carbone 3′ d’un nucléotide déjà présent dans la chaîne. Par conséquent, l’ADN ne peut être synthétisé que dans le sens 5′ vers 3′, tout en copiant un brin parent fonctionnant dans le sens 3′ vers 5′.
Chaque brin d’ADN a deux extrémités. L’extrémité 5′ de l’ADN est celle qui comporte le groupe phosphate terminal sur le carbone 5′ du désoxyribose ; l’extrémité 3′ est celle qui comporte un groupe hydroxyle (OH) terminal sur le désoxyribose du carbone 3′ du désoxyribose (voir figure \(\PageIndex{8}\)). Les deux brins sont antiparallèles, c’est-à-dire qu’ils vont dans des directions opposées. Par conséquent, l’un des brins parentaux, celui qui va de 3′ à 5′ et que l’on appelle le brin principal, peut être copié directement sur toute sa longueur (voir figure \(\PageIndex{9}\)). Cependant, l’autre brin parent – celui qui va de 5′ à 3′ et que l’on appelle le brin retardé – doit être copié de manière discontinue en courts fragments (fragments d’Okazaki) d’environ 100 à 1000 nucléotides chacun, au fur et à mesure que l’ADN se déroule. Cela se produit, comme mentionné ci-dessus, au niveau du réplisome. Le brin d’ADN en retard se détache du brin de tête, ce qui permet au réplisome de se déplacer le long des deux brins et de tirer l’ADN au fur et à mesure de la réplication. C’est l’ADN proprement dit, et non l’ADN polymérase, qui se déplace au cours de la réplication de l’ADN bactérien (voir figure \(\PageIndex{5}\)).
En outre, les enzymes ADN polymérases ne peuvent pas commencer une nouvelle chaîne d’ADN à partir de zéro. Elles peuvent seulement fixer de nouveaux nucléotides sur le groupe 3′ OH d’un nucléotide dans un brin préexistant. Par conséquent, pour commencer la synthèse du brin de tête et de chaque fragment d’ADN du brin de queue, un complexe d’ARN polymérase appelé primase est nécessaire. La primase, qui est capable de joindre des nucléotides d’ARN sans nécessiter un brin d’acide nucléique préexistant, ajoute d’abord plusieurs nucléotides d’ARN complémentaires en face des nucléotides d’ADN sur le brin principal. Cela forme ce que l’on appelle une amorce d’ARN (voir figure \(\PageIndex{10}\)).
L’ADN polymérase III remplace ensuite la primase et est capable d’ajouter des nucléotides d’ADN à l’amorce d’ARN (voir figure \(\PageIndex{11}\)). Ensuite, l’ADN polymérase II élimine l’amorce d’ARN par digestion et remplace les nucléotides d’ARN de l’amorce par les nucléotides d’ADN appropriés pour combler le vide (voir figure \(\PageIndex{12}\)). Enfin, les fragments d’ADN eux-mêmes sont reliés entre eux par l’enzyme ADN ligase (voir illustration \(\PageIndex{9}\)). Pourtant, même avec cette procédure compliquée, une macromolécule de 1000 micromètres de long d’ADN superenroulé et serré peut fabriquer une copie exacte d’elle-même en seulement 10 minutes de temps environ dans des conditions optimales, en insérant des nucléotides à un rythme d’environ 1000 nucléotides par seconde !
Animation de la réplication de l’ADN.
Courtesy of HHMI’s Biointeractive.
.