Références >> Guide de conception d’amorces pour la PCR
PCR Primer Design Guidelines
PCR (Polymerase Chain Reaction)
La réaction en chaîne par polymérase est largement tenue pour l’une des plus importantes inventions du 20e siècle en biologie moléculaire. De petites quantités de matériel génétique peuvent maintenant être amplifiées pour pouvoir a identifier, manipuler l’ADN, détecter les organismes infectieux, y compris les virus qui causent le SIDA, l’hépatite, la tuberculose, détecter les variations génétiques, y compris les mutations, dans les gènes humains et de nombreuses autres tâches.
La PCR implique les trois étapes suivantes : Dénaturation, Recuit et Extension. Tout d’abord, le matériel génétique est dénaturé, convertissant les molécules d’ADN à double brin en brins simples. Les amorces sont ensuite recuites aux régions complémentaires des molécules simple brin. Dans la troisième étape, elles sont allongées par l’action de l’ADN polymérase. Toutes ces étapes sont sensibles à la température et le choix courant des températures est respectivement de 94oC, 60oC et 70oC. Une bonne conception des amorces est essentielle à la réussite des réactions. Les considérations importantes sur la conception décrites ci-dessous sont la clé d’une amplification spécifique avec un rendement élevé. Les valeurs préférentielles indiquées sont intégrées par défaut dans tous nos produits.
1. Longueur de l’amorce : Il est généralement admis que la longueur optimale des amorces PCR est de 18 à 22 pb. Cette longueur est suffisamment longue pour une spécificité adéquate et suffisamment courte pour que les amorces se lient facilement à la matrice à la température d’hybridation.
2. Température de fusion des amorces : La température de fusion des amorces (Tm) est par définition la température à laquelle une moitié du duplex d’ADN se dissocie pour devenir simple brin et indique la stabilité du duplex. Les amorces dont la température de fusion se situe entre 52 et 58 oC donnent généralement les meilleurs résultats. Les amorces dont la température de fusion est supérieure à 65oC ont une tendance à la recombinaison secondaire. Le contenu GC de la séquence donne une bonne indication du Tm de l’amorce. Tous nos produits la calculent en utilisant la théorie thermodynamique du plus proche voisin, acceptée comme une méthode bien supérieure pour l’estimer, qui est considérée comme la plus récente et la meilleure disponible.
Formule pour le calcul de la Tm des amorces :
Température de fusion Tm(K)={ΔH/ ΔS + R ln(C)}, Ou température de fusion Tm(oC) = {ΔH/ ΔS + R ln(C)}. – 273,15 où
ΔH (kcal/mole) : H est l’enthalpie. L’enthalpie est la quantité d’énergie thermique possédée par les substances. ΔH est la variation de l’enthalpie. Dans la formule ci-dessus, le ΔH est obtenu en additionnant toutes les valeurs d’enthalpie des paires de di-nucléotides de chaque paire de bases voisines les plus proches.
ΔS (kcal/mole) : S est la quantité de désordre que présente un système est appelée entropie. ΔS est la variation de l’entropie. Ici, il est obtenu en additionnant toutes les valeurs d’entropie des paires de di-nucléotides de chaque paire de bases voisines les plus proches. Une correction de sel supplémentaire est ajoutée car les paramètres de plus proche voisin ont été obtenus à partir d’études de fusion de l’ADN menées dans un tampon Na+ 1M et c’est la condition par défaut utilisée pour tous les calculs.
ΔS (correction de sel) = ΔS (1M NaCl )+ 0.368 x N x ln()
Où
N est le nombre de paires de nucléotides dans l’amorce ( longueur de l’amorce -1).
est l’équivalent en sel en mM.
calcul:
= concentration en ions monovalents +4 x Mg2+ libre.
3. température de recuit de l’amorce : La température de fusion de l’amorce est l’estimation de la stabilité de l’hybride ADN-ADN et critique pour déterminer la température de recuit. Une Ta trop élevée produira une hybridation amorce-template insuffisante, ce qui entraînera un faible rendement en produits PCR. Une Ta trop basse peut éventuellement conduire à des produits non spécifiques causés par un nombre élevé de mésappariements de paires de bases. On constate que la tolérance aux mésappariements a la plus forte influence sur la spécificité de la PCR.
Ta = 0,3 x Tm(amorce) + 0,7 Tm (produit) – 14.9
où,
Tm(amorce) = Température de fusion des amorces
Tm(produit) = Température de fusion du produit
4. Contenu GC : Le contenu GC (le nombre de G et de C dans l’amorce en pourcentage du total des bases) de l’amorce doit être de 40-60%.
5. Pince GC : La présence de bases G ou C dans les cinq dernières bases de l’extrémité 3′ des amorces (GC clamp) permet de favoriser la liaison spécifique à l’extrémité 3′ en raison de la liaison plus forte des bases G et C. Il faut éviter plus de 3 G ou C dans les 5 dernières bases de l’extrémité 3′ de l’amorce.
6. Structures secondaires de l’amorce : La présence des structures secondaires de l’amorce produites par des interactions intermoléculaires ou intramoléculaires peut conduire à un rendement faible ou nul du produit. Elles affectent négativement le recuit de la matrice d’amorce et donc l’amplification. Elles réduisent considérablement la disponibilité des amorces pour la réaction.
i) Les épingles à cheveux : Elles sont formées par une interaction intramoléculaire au sein de l’amorce et doivent être évitées. De manière optimale, on tolère généralement une épingle à cheveux à l’extrémité 3′ avec un ΔG de -2 kcal/mol et une épingle à cheveux interne avec un ΔG de -3 kcal/mol.
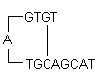
Définition du ΔG : L’énergie libre de Gibbs G est la mesure de la quantité de travail qui peut être extraite d’un processus fonctionnant à une pression constante. C’est la mesure de la spontanéité de la réaction. La stabilité d’une épingle à cheveux est communément représentée par sa valeur ΔG, l’énergie requise pour briser la structure secondaire. Une valeur négative plus grande pour le ΔG indique des épingles à cheveux stables et indésirables. La présence d’épingles à cheveux à l’extrémité 3′ affecte le plus négativement la réaction.
ΔG = ΔH – TΔS
ii) Autodimère : Un autodimère d’amorce est formé par des interactions intermoléculaires entre les deux amorces (même sens), où l’amorce est homologue à elle-même. En général, une grande quantité d’amorces est utilisée dans la PCR par rapport à la quantité de gène cible. Lorsque les amorces forment des dimères intermoléculaires beaucoup plus facilement qu’elles ne s’hybrident à l’ADN cible, elles réduisent le rendement du produit. De manière optimale, un auto-dimère d’extrémité 3′ avec un ΔG de -5 kcal/mol et un auto-dimère interne avec un ΔG de -6 kcal/mol sont généralement tolérés.
iii) Dimère croisé : Les dimères croisés d’amorces sont formés par l’interaction intermoléculaire entre les amorces sens et antisens, lorsqu’elles sont homologues. De manière optimale, un dimère croisé à l’extrémité 3′ avec un ΔG de -5 kcal/mol et un dimère croisé interne avec un ΔG de -6 kcal/mol sont tolérés généralement.
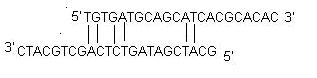
7. répétitions : Une répétition est un di-nucléotide se produisant de nombreuses fois consécutivement et doit être évitée parce qu’ils peuvent se tromper de priorité. Par exemple : ATATATAT. Un nombre maximum de répétitions de di-nucléotides acceptable dans un oligo est de 4 di-nucléotides.
8. Courses : Les amorces avec de longues courses d’une seule base doivent généralement être évitées car elles peuvent se tromper d’ordre. Par exemple, AGCGGGGGATGGGG a des runs de base ‘G’ de valeur 5 et 4. Un nombre maximum de parcours accepté est de 4bp.
9. Stabilité de l’extrémité 3′ : C’est la valeur ΔG maximale des cinq bases de l’extrémité 3′. Une extrémité 3′ instable (ΔG moins négatif) entraînera moins de faux amorçage.
10. Éviter la structure secondaire du modèle : Une séquence d’acide nucléique simple brin est très instable et se replie en conformations (structures secondaires). La stabilité de ces structures secondaires de template dépend largement de leur énergie libre et de leurs températures de fusion(Tm). La prise en compte des structures secondaires de la matrice est importante pour la conception des amorces, en particulier dans le cadre de la qPCR. Si les amorces sont conçues sur une structure secondaire qui est stable même au-dessus des températures d’hybridation, les amorces sont incapables de se lier à la matrice et le rendement du produit PCR est considérablement affecté. Il est donc important de concevoir des amorces dans les régions des matrices qui ne forment pas de structures secondaires stables pendant la réaction PCR. Nos produits déterminent les structures secondaires de la matrice et conçoivent des amorces les évitant.
11. Éviter l’homologie croisée : Pour améliorer la spécificité des amorces, il est nécessaire d’éviter les régions d’homologie. Les amorces conçues pour une séquence ne doivent pas amplifier d’autres gènes dans le mélange. En général, les amorces sont conçues puis testées par BLAST pour vérifier leur spécificité. Nos produits offrent une meilleure alternative. Vous pouvez éviter les régions d’homologie croisée lors de la conception des amorces. Vous pouvez effectuer un BLAST des modèles par rapport à la base de données non redondante appropriée et le logiciel interprétera les résultats. Il identifiera les régions d’homologies croisées significatives dans chaque modèle et les évitera pendant la recherche d’amorces.
Paramètres pour la conception de paires d’amorces
1. Longueur de l’amplicon : La longueur de l’amplicon est dictée par les objectifs expérimentaux. Pour la qPCR, la longueur cible est plus proche de 100 pb et pour la PCR standard, elle est proche de 500 pb. Si vous connaissez les positions de chaque amorce par rapport à la matrice, le produit est calculé comme suit : Longueur du produit = (Position de l’amorce antisens-Position de l’amorce sens) + 1.
2. Position du produit : L’amorce peut être située près de l’extrémité 5′, de l’extrémité 3′ ou n’importe où dans la longueur spécifiée. Généralement, la séquence proche de l’extrémité 3′ est connue avec plus de confiance et donc préférée le plus souvent.
3. Tm du produit : La température de fusion (Tm) est la température à laquelle une moitié du duplex d’ADN se dissocie et devient simple brin. La stabilité du duplex ADN amorce-template peut être mesurée par la température de fusion (Tm).
4. Température de recuit optimale (Ta Opt) : La formule de Rychlik est la plus respectée. Nos produits utilisent cette formule pour la calculer et des milliers de nos clients ont rapporté de bons résultats en l’utilisant pour l’étape de recuit du cycle PCR. Elle permet généralement d’obtenir un bon rendement en produits PCR avec une production minimale de faux produits.
Ta Opt = 0,3 x(Tm de l’amorce) + 0,7 x(Tm du produit) – 14.9
où
Tm de l’amorce est la température de fusion de la paire amorce-template moins stable
Tm du produit est la température de fusion du produit PCR.
5. Calcul de l’inadéquation de la Tm de la paire d’amorce : Les deux amorces d’une paire d’amorces doivent avoir des températures de fusion étroitement appariées pour maximiser le rendement du produit PCR. La différence de 5oC ou plus peut conduire à aucune amplification.
Conception d’amorce à l’aide de logiciels
Il existe un certain nombre d’outils de conception d’amorce qui peuvent aider à la conception d’amorce PCR pour les nouveaux utilisateurs et les utilisateurs expérimentés. Ces outils peuvent réduire le coût et le temps impliqués dans l’expérimentation en diminuant les chances d’échec de l’expérimentation.
Primer Premier suit toutes les directives spécifiées pour la conception d’amorces PCR. Primer Premier peut être utilisé pour concevoir des amorces pour les modèles uniques, les alignements, la conception d’amorces dégénérées, l’analyse des enzymes de restriction. l’analyse des contigs et la conception d’amorces de séquençage.
Les directives pour la conception d’amorces de qPCR varient légèrement. Des logiciels tels qu’AlleleID et Beacon Designer peuvent concevoir des amorces et des sondes oligonucléotidiques pour des tests de détection complexes tels que les tests multiplex, la conception d’amorces inter-espèces, la conception d’amorces spécifiques à une espèce et la conception d’amorces pour réduire le coût de l’expérimentation.
PrimerPlex est un logiciel qui peut concevoir des amorces pour la PCR multiplex et les tests de génotypage SNP multiplex.
.