Hivatkozások >> PCR-primerek
PCR-primer tervezési útmutató
PCR (polimeráz láncreakció)
A polimeráz láncreakciót széles körben a 20. század egyik legfontosabb molekuláris biológiai találmányának tartják. A genetikai anyag kis mennyiségeit ma már fel lehet sokszorozni, hogy képesek legyünk a DNS azonosítására, manipulálására, fertőző organizmusok kimutatására, beleértve az AIDS-et, hepatitiszt, tuberkulózist okozó vírusokat, az emberi gének genetikai variációinak, beleértve a mutációkat is, kimutatására és számos más feladatra.
A PCR a következő három lépést foglalja magában: Denaturálás, lágyítás és hosszabbítás. Először a genetikai anyagot denaturálják, a kettős szálú DNS-molekulákat egyszálúvá alakítják. Ezután a primereket az egyszálú molekulák komplementer régióihoz lágyítják. A harmadik lépésben a DNS-polimeráz hatására meghosszabbítják őket. Mindezek a lépések hőmérséklet-érzékenyek, és az általánosan választott hőmérséklet 94oC, 60oC és 70oC. A sikeres reakciókhoz elengedhetetlen a jó primertervezés. Az alábbiakban ismertetett fontos tervezési megfontolások kulcsfontosságúak a nagy hozamú specifikus amplifikációhoz. A feltüntetett preferált értékek alapértelmezés szerint minden termékünkbe be vannak építve.
1. Primer hossza: Általánosan elfogadott, hogy a PCR-primerek optimális hossza 18-22 bp. Ez a hossz elég hosszú a megfelelő specificitáshoz, és elég rövid ahhoz, hogy a primerek a lágyulási hőmérsékleten könnyen kötődjenek a templáthoz.
2. Primer olvadási hőmérséklet: A primer olvadási hőmérséklete (Tm) definíció szerint az a hőmérséklet, amelyen a DNS-duplex egyik fele disszociál és egyszálúvá válik, és jelzi a duplex stabilitását. Az 52-58 oC közötti olvadási hőmérsékletű primerek általában a legjobb eredményeket adják. A 65oC feletti olvadási hőmérsékletű primerek hajlamosak a másodlagos lágyulásra. A szekvencia GC-tartalma jól jelzi a primer Tm-jét. Minden termékünk a legközelebbi szomszédos termodinamikai elmélet alapján számítja ki, amelyet sokkal jobb becslési módszerként fogadnak el, és amelyet a legújabb és legjobb elérhetőnek tartanak.
A primer Tm kiszámításának képlete:
Az olvadási hőmérséklet Tm(K)={ΔH/ ΔS + R ln(C)}, Vagy olvadási hőmérséklet Tm(oC) = {ΔH/ ΔS + R ln(C)} – 273,15 ahol
ΔH (kcal/mol) : H az Enthalpia. Az Enthalpia az anyagok által birtokolt hőenergia mennyisége. ΔH az Enthalpia változása. A fenti képletben a ΔH-t az egyes legközelebbi szomszédos bázispárok összes di-nukleotidpár entalpiaértékének összeadásával kapjuk.
ΔS (kcal/mol) : Az S a rendszer által mutatott rendezetlenség mértékét nevezzük entrópiának. ΔS az entrópia változása. Itt úgy kapjuk meg, hogy összeadjuk az összes di-nukleotidpár entrópiaértékét az egyes legközelebbi szomszédos bázispárokhoz. Egy további sókorrekciót adunk hozzá, mivel a legközelebbi szomszéd paramétereket 1M Na+ pufferben végzett DNS-olvadási vizsgálatokból kaptuk, és ez az összes számításhoz használt alapértelmezett feltétel.
ΔS (sókorrekció) = ΔS (1M NaCl )+ 0.368 x N x ln()
Hol
N a primerben lévő nukleotidpárok száma ( primer hossza -1).
a sóegyenérték mM-ben.
számítás:
= Monovalens ion koncentráció +4 x szabad Mg2+.
3. Primer izzítási hőmérséklet: A primer olvadási hőmérséklete a DNS-DNS hibrid stabilitásának becslése, és kritikus a lágyítási hőmérséklet meghatározásához. A túl magas Ta elégtelen primer-sablon hibridizációt eredményez, ami alacsony PCR-termékhozamot eredményez. A túl alacsony Ta esetleg a bázispár-eltérések nagy száma miatt nem specifikus termékekhez vezethet. Úgy találták, hogy a hibatűrésnek van a legerősebb hatása a PCR-specifikusságra.
Ta = 0,3 x Tm(primer) + 0,7 Tm (termék) – 14.9
hol,
Tm(primer) = a primerek olvadási hőmérséklete
Tm(termék) = a termék olvadási hőmérséklete
4. GC tartalom: A primer GC-tartalmának (a primerben lévő G-k és C-k száma az összes bázis százalékában) 40-60%-nak kell lennie.
5. GC-megszorítás: A G vagy C bázisok jelenléte a primerek 3′ végétől számított utolsó öt bázison belül (GC clamp) elősegíti a specifikus kötődést a 3′ végén a G és C bázisok erősebb kötődése miatt. A primer 3′ végének utolsó 5 bázisában 3-nál több G vagy C bázis kerülendő.
6. Primer másodlagos szerkezetek: Az intermolekuláris vagy intramolekuláris kölcsönhatások által létrehozott primer másodlagos struktúrák jelenléte a termék gyenge vagy semmilyen hozamához nem vezethet. Ezek kedvezőtlenül befolyásolják a primer templát lágyulását és ezáltal az amplifikációt. Nagymértékben csökkentik a primerek hozzáférhetőségét a reakcióhoz.
i) Hajszálerek: A primeren belüli intramolekuláris kölcsönhatás révén jön létre, és kerülni kell. Optimális esetben a 3′ végi hajtű -2 kcal/mol ΔG-vel és a belső hajtű -3 kcal/mol ΔG-vel általában tolerálható.
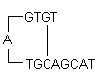
ΔG definíció: A G Gibbs-féle szabad energia G annak a munkának a mértéke, amely egy állandó nyomáson működő folyamatból kivonható. Ez a reakció spontaneitásának a mértéke. A hajtű stabilitását általában a ΔG értékével, a másodlagos szerkezet felbomlásához szükséges energiával szokták ábrázolni. A ΔG nagyobb negatív értéke stabil, nemkívánatos hajszálpontokat jelez. A hajszálerek jelenléte a 3′-végen befolyásolja legkedvezőtlenebbül a reakciót.
ΔG = ΔH – TΔS
ii) Öndimer: A primer öndimer a két (azonos értelemben vett) primer közötti intermolekuláris kölcsönhatások révén jön létre, ahol a primer önmagával homológ. Általában a PCR során a célgén mennyiségéhez képest nagy mennyiségű primert használunk. Amikor a primerek sokkal könnyebben képeznek intermolekuláris dimereket, mint a cél-DNS-hez hibridizálódva, csökkentik a termékhozamot. Optimális esetben a 3′ végi öndimer -5 kcal/mol ΔG-vel és a belső öndimer -6 kcal/mol ΔG-vel általában tolerálható.
iii) Keresztdimer: A primer keresztdimerek a sense és antisense primerek közötti intermolekuláris kölcsönhatás során jönnek létre, ahol homológok. Optimális esetben egy 3′ végi keresztdimer -5 kcal/mol ΔG-vel és egy belső keresztdimer -6 kcal/mol ΔG-vel általában tolerálható.
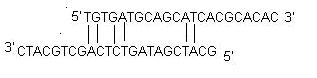
7. Ismétlések: Az ismétlődés olyan di-nukleotid, amely többször egymás után fordul elő, és kerülendő, mert félreprimálódhatnak. Például: ATATATATAT. A di-nukleotid ismétlődések maximálisan elfogadható száma egy oligóban 4 di-nukleotid.
8. Futások: A hosszú, egyetlen bázisból álló futásokat tartalmazó primereket általában kerülni kell, mivel félreprime-olhatnak. Az AGCGGGGGGGATGGGGGG például 5 és 4 értékű “G” bázist tartalmaz. A maximálisan elfogadott futások száma 4bp.
9. A 3′ vég stabilitása: Ez a 3′ végétől számított öt bázis maximális ΔG-értéke. Egy instabil 3′ vég (kisebb negatív ΔG) kevesebb hamis priminget eredményez.
10. Kerülje a sablon másodlagos szerkezetét: Az egyszálú nukleinsav szekvenciák rendkívül instabilak és konformációkba (másodlagos struktúrákba) hajtogatódnak. Ezeknek a sablon másodlagos struktúráknak a stabilitása nagymértékben függ a szabad energiától és az olvadási hőmérséklettől(Tm). A templát másodlagos struktúrák figyelembevétele fontos a primerek tervezésénél, különösen a qPCR-ben. Ha a primereket olyan másodlagos struktúrákra tervezik, amelyek még a lágyulási hőmérséklet felett is stabilak, a primerek nem tudnak kötődni a templáthoz, és a PCR-termék hozama jelentősen csökken. Ezért fontos, hogy a primereket a templátok azon régióiba tervezzük, amelyek a PCR-reakció során nem képeznek stabil másodlagos struktúrákat. Termékeink meghatározzák a templát másodlagos szerkezeteit, és olyan primereket terveznek, amelyek elkerülik azokat.
11. Kereszt-homológia elkerülése: A primerek specificitásának javítása érdekében el kell kerülni a homológia régióit. Az egy szekvenciára tervezett primerek nem amplifikálhatnak más géneket a keverékben. Általában primereket terveznek, majd BLAST-olnak a specificitás tesztelésére. Termékeink jobb alternatívát kínálnak. A primerek tervezése során elkerülheti a kereszthomológiás régiókat. A sablonokat BLAST-olhatja a megfelelő nem redundáns adatbázissal szemben, és a szoftver értelmezi az eredményeket. Azonosítja a jelentős kereszthomológiájú régiókat az egyes sablonokban, és elkerüli azokat a primerkeresés során.
Paraméterek a primerpárok tervezéséhez
1. Amplikon hossza: Az amplikon hosszát a kísérleti célok diktálják. A qPCR esetében a célhossz közelebb van a 100 bp-hoz, a standard PCR esetében pedig 500 bp közelében van. Ha ismerjük az egyes primerek pozícióját a templáthoz képest, a termék kiszámítása a következőképpen történik: A termék hossza = (az antisense primer pozíciója – az sense primer pozíciója) + 1.
2. A termék pozíciója: A primer elhelyezkedhet az 5′ vég közelében, a 3′ végen vagy a megadott hosszon belül bárhol. Általában a 3′ véghez közeli szekvencia nagyobb biztonsággal ismert, ezért azt részesítik előnyben a leggyakrabban.
3. A termék Tm-je: Az olvadási hőmérséklet (Tm) az a hőmérséklet, amelyen a DNS-duplex egyik fele disszociál és egyszálúvá válik. A primer-minta DNS-duplex stabilitása az olvadási hőmérséklet (Tm) alapján mérhető.
4. Optimális izzítási hőmérséklet (Ta Opt): A Rychlik-féle képletet tartjuk leginkább tiszteletben. Termékeink ezt a képletet használják a kiszámításához, és ügyfeleink ezrei számoltak be jó eredményekről, amikor ezt használták a PCR-ciklus lágyítási lépésére. Általában jó PCR-termékhozamot eredményez, minimális hamis termék keletkezése mellett.
Ta Opt = 0,3 x(primer Tm) + 0,7 x(termék Tm) – 14.9
hol
A primer Tm-je a kevésbé stabil primer-sablon pár olvadási hőmérséklete
A termék Tm-je a PCR termék olvadási hőmérséklete.
5. Primerpár Tm Mismatch számítás: A PCR-termékhozam maximalizálása érdekében a primerpár két primerének közel azonos olvadási hőmérsékletűnek kell lennie. Az 5oC vagy annál nagyobb különbség nem vezethet amplifikációhoz.
Primertervezés szoftverek segítségével
Egy sor primertervezési eszköz áll rendelkezésre, amelyek segítséget nyújthatnak a PCR-primerek tervezésében az új és a tapasztalt felhasználók számára egyaránt. Ezek az eszközök csökkenthetik a kísérletezéssel járó költségeket és időt, csökkentve a sikertelen kísérletezés esélyét.
A Primer Premier a PCR-primerek tervezésére meghatározott összes irányelvet követi. A Primer Premier használható primerek tervezéséhez egyedi templátokhoz, igazításokhoz, degenerált primerek tervezéséhez, restrikciós enzim analízishez. contig analízishez és szekvenáló primerek tervezéséhez.
A qPCR primerek tervezésére vonatkozó irányelvek némileg eltérnek. Az olyan szoftverek, mint az AlleleID és a Beacon Designer képesek primerek és oligonukleotid szondák tervezésére komplex kimutatási próbákhoz, például multiplex próbákhoz, fajok közötti primertervezéshez, fajspecifikus primertervezéshez és primertervezéshez a kísérleti költségek csökkentése érdekében.
A PrimerPlex egy olyan szoftver, amely képes primerek tervezésére multiplex PCR és multiplex SNP genotipizálási próbákhoz.