B. Replicazione del DNA nei batteri
In generale, il DNA si replica mediante lo srotolamento dell’elica, la separazione dei filamenti mediante la rottura dei legami idrogeno tra i filamenti complementari, e la sintesi di due nuovi filamenti mediante l’accoppiamento di basi complementari. La replicazione inizia in un sito specifico del DNA chiamato origine della replicazione (oriC).
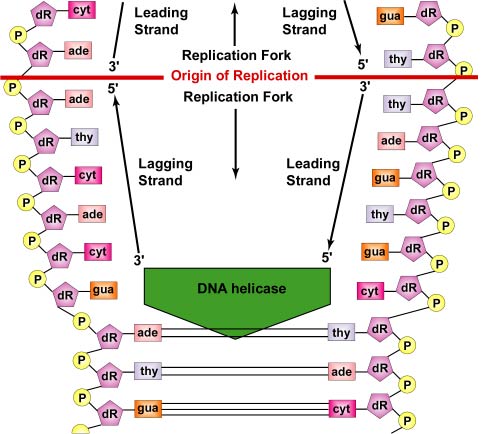
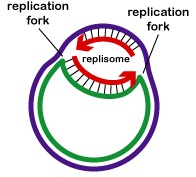
La replicazione del DNA è bidirezionale dall’origine della replicazione. Per iniziare la replicazione del DNA, gli enzimi di svolgimento chiamati elicasi del DNA fanno sì che brevi segmenti dei due filamenti di DNA dei genitori si srotolino e si separino l’uno dall’altro all’origine della replicazione per formare due forchette di replicazione a forma di “Y”. Queste forchette di replicazione sono il sito effettivo della copia del DNA (Figura \PageIndex{3}\). Tutte le proteine coinvolte nella replicazione del DNA si aggregano alle forchette di replicazione per formare un complesso di replicazione chiamato replisoma (Figura \PageIndex{4}).
Le proteine leganti il singolo filamento si legano alle regioni a singolo filamento in modo che i due filamenti non si riuniscano. Lo srotolamento dell’elica a doppio filamento genera delle supercoil positive davanti alla forcella di replicazione. Gli enzimi chiamati topoisomerasi contrastano questo fenomeno producendo rotture nel DNA e poi le ricongiungono per formare supercoil negative, in modo da alleviare questo stress nella molecola elicoidale durante la replicazione.
Come i filamenti continuano a srotolarsi e a separarsi in entrambe le direzioni intorno all’intera molecola di DNA, nuovi filamenti complementari sono prodotti dal legame a idrogeno dei nucleotidi liberi del DNA con quelli di ciascun filamento genitore. Quando i nuovi nucleotidi si allineano di fronte a ciascun filamento genitore per legame a idrogeno, gli enzimi chiamati DNA polimerasi uniscono i nucleotidi per mezzo di legami fosfodiesteri. In realtà, i nucleotidi che si allineano per accoppiamento di basi complementari sono trifosfati deossinucleotidici, composti da una base azotata, deossiribosio, e tre fosfati. Quando il legame fosfodiestere si forma tra il gruppo fosfato 5′ del nuovo nucleotide e l’OH 3′ dell’ultimo nucleotide nel filamento di DNA, due dei fosfati vengono rimossi fornendo energia per il legame (vedi figura \(\PageIndex{6}). Alla fine, ogni filamento genitore serve come modello per sintetizzare una copia complementare di se stesso, dando luogo alla formazione di due molecole di DNA identiche (vedi Figura \(\PageIndex{7}}). Nei batteri, le proteine Par funzionano per separare i cromosomi batterici ai poli opposti della cellula durante la divisione cellulare. Si legano all’origine della replicazione del DNA e tirano o spingono fisicamente i cromosomi a parte, in modo simile all’apparato mitotico delle cellule eucariotiche. Le proteine Fts, come FtsK nel divisoma, aiutano anche a separare il cromosoma batterico replicato.
GIF animazione che illustra la replicazione del DNA per accoppiamento di basi complementari
In realtà, la replicazione del DNA è più complicata di così a causa della natura delle DNA polimerasi. Gli enzimi della DNA polimerasi sono solo in grado di unire il gruppo fosfato al carbonio 5′ di un nuovo nucleotide al gruppo idrossile (OH) del carbonio 3′ di un nucleotide già presente nella catena. Di conseguenza, il DNA può essere sintetizzato solo in direzione da 5′ a 3′ copiando un filamento genitore che corre in direzione da 3′ a 5′.
Ogni filamento di DNA ha due estremità. L’estremità 5′ del DNA è quella con il gruppo fosfato terminale sul carbonio 5′ del desossiribosio; l’estremità 3′ è quella con un gruppo idrossile (OH) terminale sul desossiribosio del carbonio 3′ del desossiribosio (vedi figura \(\PageIndex{8}). I due filamenti sono antiparalleli, cioè corrono in direzioni opposte. Pertanto, un filamento genitore – quello che corre da 3′ a 5′ e chiamato filamento principale – può essere copiato direttamente per tutta la sua lunghezza (vedi Figura \(\PageIndex{9}). Tuttavia, l’altro filamento genitore – quello che corre da 5′ a 3′ e chiamato filamento ritardatario – deve essere copiato in modo discontinuo in brevi frammenti (frammenti Okazaki) di circa 100-1000 nucleotidi ciascuno mentre il DNA si srotola. Questo avviene, come detto sopra, nel replisoma. Il filamento di DNA ritardatario si stacca dal filamento principale e questo permette al replisoma di muoversi lungo entrambi i filamenti e di far passare il DNA durante la replicazione. È il DNA vero e proprio, non la DNA polimerasi, che si muove durante la replicazione del DNA batterico (vedi Figura \PageIndex{5}).
Inoltre, gli enzimi della DNA polimerasi non possono iniziare una nuova catena di DNA da zero. Possono solo attaccare nuovi nucleotidi sul gruppo 3′ OH di un nucleotide in un filamento preesistente. Pertanto, per iniziare la sintesi del filamento principale e di ogni frammento di DNA del filamento ritardatario, è necessario un complesso di RNA polimerasi chiamato primasi. La primasi, che è in grado di unire nucleotidi di RNA senza richiedere un filamento preesistente di acido nucleico, aggiunge prima diversi nucleotidi di RNA complementari di fronte ai nucleotidi di DNA sul filamento principale. Questo forma quello che viene chiamato un primer RNA (vedi Figura \PageIndex{10}).
La DNA polimerasi III sostituisce poi la primasi ed è in grado di aggiungere nucleotidi di DNA al primer RNA (vedi Figura \PageIndex{11}). Successivamente, la DNA polimerasi II digerisce il primer RNA e sostituisce i nucleotidi RNA del primer con i nucleotidi di DNA appropriati per riempire il vuoto (vedi figura \(\PageIndex{12}). Infine, i frammenti di DNA stessi sono agganciati insieme dall’enzima DNA ligasi (vedi Figura \(\PageIndex{9})). Eppure, anche con questa complicata procedura, una macromolecola lunga 1000 micrometri di DNA strettamente impacchettato e superavvolto può fare una copia esatta di se stessa in soli 10 minuti di tempo in condizioni ottimali, inserendo nucleotidi ad una velocità di circa 1000 nucleotidi al secondo!
Animazione della replicazione del DNA.
Courtesy of HHMI’s Biointeractive.