References >> PCR Primer
PCR Primer Design Guidelines
PCR (Polymerase Chain Reaction)
Polymerase Chain Reaction è ampiamente considerata come una delle più importanti invenzioni del 20° secolo in biologia molecolare. Piccole quantità di materiale genetico possono ora essere amplificate per essere in grado di identificare, manipolare il DNA, rilevare organismi infettivi, compresi i virus che causano l’AIDS, l’epatite, la tubercolosi, rilevare variazioni genetiche, comprese le mutazioni, nei geni umani e numerosi altri compiti.
PCR comporta i seguenti tre passaggi: Denaturazione, Ricottura ed Estensione. In primo luogo, il materiale genetico viene denaturato, convertendo le molecole di DNA a doppio filamento in filamenti singoli. I primer vengono poi ricotti alle regioni complementari delle molecole a singolo filamento. Nel terzo passo, vengono estesi dall’azione della DNA polimerasi. Tutti questi passaggi sono sensibili alla temperatura e la scelta comune delle temperature è 94oC, 60oC e 70oC rispettivamente. Una buona progettazione dei primer è essenziale per il successo delle reazioni. Le importanti considerazioni di progettazione descritte di seguito sono la chiave per un’amplificazione specifica ad alta resa. I valori preferiti indicati sono incorporati di default in tutti i nostri prodotti.
1. Lunghezza del primer: È generalmente accettato che la lunghezza ottimale dei primer PCR è di 18-22 bp. Questa lunghezza è abbastanza lunga per un’adeguata specificità e abbastanza corta perché i primer si leghino facilmente al modello alla temperatura di annealing.
2. Temperatura di fusione dei primer: La temperatura di fusione dei primer (Tm) per definizione è la temperatura alla quale una metà del duplex del DNA si dissocia per diventare a singolo filamento e indica la stabilità del duplex. I primer con temperature di fusione nell’intervallo di 52-58 oC generalmente producono i migliori risultati. I primer con temperature di fusione superiori a 65oC hanno una tendenza all’annealing secondario. Il contenuto di GC della sequenza dà una discreta indicazione della Tm del primer. Tutti i nostri prodotti la calcolano usando la teoria termodinamica del vicino più vicino, accettata come un metodo molto superiore per stimarla, che è considerato il più recente e migliore disponibile.
Formula per il calcolo della Tm del primer:
Temperatura di fusione Tm(K)={ΔH/ ΔS + R ln(C)}, o Temperatura di fusione Tm(oC) = {ΔH/ ΔS + R ln(C)} – 273.15 dove
ΔH (kcal/mole) : H è l’Entalpia. L’entalpia è la quantità di energia termica posseduta dalle sostanze. ΔH è la variazione dell’entalpia. Nella formula precedente il ΔH si ottiene sommando tutti i valori di entalpia delle coppie di di-nucleotidi di ogni coppia di basi vicine.
ΔS (kcal/mole): S è la quantità di disordine che un sistema esibisce, chiamata entropia. ΔS è il cambiamento dell’entropia. Qui si ottiene sommando tutti i valori di entropia delle coppie di-nucleotidi di ogni coppia di basi vicine. Viene aggiunta un’ulteriore correzione salina in quanto i parametri del Nearest Neighbor sono stati ottenuti da studi di fusione del DNA condotti in un buffer 1M Na+ e questa è la condizione di default utilizzata per tutti i calcoli.
ΔS (correzione salina) = ΔS (1M NaCl )+ 0.368 x N x ln()
Dove
N è il numero di coppie di nucleotidi nel primer (lunghezza del primer -1).
è il sale equivalente in mM.
calcolo:
= concentrazione di ioni monovalenti +4 x Mg2+ libero.
3. Temperatura di ricottura del primer: La temperatura di fusione del primer è la stima della stabilità dell’ibrido DNA-DNA ed è fondamentale per determinare la temperatura di annealing. Una Ta troppo alta produrrà un’ibridazione primer-template insufficiente con conseguente bassa resa del prodotto PCR. Una Ta troppo bassa può eventualmente portare a prodotti non specifici causati da un alto numero di coppie di basi non corrispondenti. La tolleranza ai mismatch è risultata avere la più forte influenza sulla specificità della PCR.
Ta = 0,3 x Tm(primer) + 0,7 Tm (prodotto) – 14.9
dove,
Tm(primer) = temperatura di fusione dei primer
Tm(prodotto) = temperatura di fusione del prodotto
4. Contenuto di GC: Il contenuto di GC (il numero di G e C nel primer come percentuale delle basi totali) del primer dovrebbe essere 40-60%.
5. GC Clamp: La presenza di basi G o C entro le ultime cinque basi dall’estremità 3′ dei primer (GC clamp) aiuta a promuovere il legame specifico all’estremità 3′ a causa del legame più forte delle basi G e C. Più di 3 G o C dovrebbero essere evitate nelle ultime 5 basi all’estremità 3′ del primer.
6. Strutture secondarie del primer: La presenza di strutture secondarie del primer prodotte da interazioni intermolecolari o intramolecolari può portare a una scarsa o nulla resa del prodotto. Influenzano negativamente l’annealing del template del primer e quindi l’amplificazione. Riducono notevolmente la disponibilità di primer per la reazione.
i) Hairpins: Si formano per interazione intramolecolare all’interno del primer e dovrebbero essere evitate. In modo ottimale, una forcina all’estremità 3′ con un ΔG di -2 kcal/mol e una forcina interna con un ΔG di -3 kcal/mol sono generalmente tollerate.
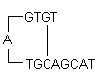
Definizione di ΔG: L’energia libera di Gibbs G è la misura della quantità di lavoro che può essere estratta da un processo operante a pressione costante. È la misura della spontaneità della reazione. La stabilità della forcina è comunemente rappresentata dal suo valore ΔG, l’energia richiesta per rompere la struttura secondaria. Un valore negativo maggiore per il ΔG indica forcine stabili e indesiderabili. La presenza di forcine all’estremità 3′ influenza più negativamente la reazione.
ΔG = ΔH – TΔS
ii) Self Dimer: Un primer self-dimer è formato da interazioni intermolecolari tra i due primer (stesso senso), dove il primer è omologo a se stesso. Generalmente nella PCR si usa una grande quantità di primer rispetto alla quantità di gene bersaglio. Quando i primer formano dimeri intermolecolari molto più facilmente dell’ibridazione con il DNA bersaglio, riducono la resa del prodotto. In genere si tollera in modo ottimale un auto dimero all’estremità 3′ con un ΔG di -5 kcal/mol e un auto dimero interno con un ΔG di -6 kcal/mol.
iii) Cross Dimer: i cross dimeri dei primer si formano per interazione intermolecolare tra primer senso e antisenso, quando sono omologhi. In modo ottimale un dimero incrociato all’estremità 3′ con un ΔG di -5 kcal/mol e un dimero incrociato interno con un ΔG di -6 kcal/mol è generalmente tollerato.
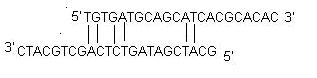
7. Ripetizioni: Una ripetizione è un di-nucleotide che si verifica molte volte consecutivamente e dovrebbe essere evitata perché può sbagliare la priorità. Per esempio: ATATATAT. Un numero massimo di ripetizioni di di-nucleotidi accettabile in un oligo è di 4 di-nucleotidi.
8. Corse: I primer con lunghe corse di una singola base dovrebbero generalmente essere evitati perché possono avere un errore di priorità. Per esempio, AGCGGGGGATGGGG ha corse di base ‘G’ di valore 5 e 4. Il numero massimo di corse accettate è 4 bp.
9. 3′ Stabilità finale: È il massimo valore ΔG delle cinque basi dell’estremità 3′. Un’estremità 3′ instabile (ΔG meno negativo) risulterà in meno falsi priming.
10. Evitare la struttura secondaria del template: Una sequenza di acido nucleico a singolo filamento è altamente instabile e si ripiega in conformazioni (strutture secondarie). La stabilità di queste strutture secondarie dipende in gran parte dalla loro energia libera e dalle temperature di fusione (Tm). La considerazione delle strutture secondarie del modello è importante nella progettazione dei primer, specialmente nella qPCR. Se i primer sono progettati su strutture secondarie che sono stabili anche al di sopra delle temperature di annealing, i primer non sono in grado di legarsi al modello e la resa del prodotto PCR è significativamente influenzata. Quindi, è importante progettare primer nelle regioni dei template che non formano strutture secondarie stabili durante la reazione di PCR. I nostri prodotti determinano le strutture secondarie del modello e progettano primer che le evitano.
11. Evitare l’omologia incrociata: Per migliorare la specificità dei primer è necessario evitare le regioni di omologia. I primer progettati per una sequenza non devono amplificare altri geni nella miscela. Comunemente, i primer sono progettati e poi BLASTati per testare la specificità. I nostri prodotti offrono un’alternativa migliore. È possibile evitare le regioni di omologia incrociata durante la progettazione dei primer. È possibile eseguire il BLAST dei modelli contro il database non ridondante appropriato e il software interpreterà i risultati. Identificherà le regioni con omologie incrociate significative in ogni modello e le eviterà durante la ricerca dei primer.
Parametri per la progettazione delle coppie di primer
1. Lunghezza dell’amplicone: La lunghezza dell’amplicone è dettata dagli obiettivi sperimentali. Per la qPCR, la lunghezza dell’obiettivo è più vicina ai 100 bp e per la PCR standard, è vicina ai 500 bp. Se conosci le posizioni di ogni primer rispetto al modello, il prodotto è calcolato come: Lunghezza del prodotto = (Posizione del primer antisenso-Posizione del primer senso) + 1.
2. Posizione del prodotto: Il primer può essere situato vicino all’estremità 5′, all’estremità 3′ o in qualsiasi punto della lunghezza specificata. Generalmente, la sequenza vicino all’estremità 3′ è conosciuta con maggiore sicurezza e quindi preferita più frequentemente.
3. Tm del prodotto: La temperatura di fusione (Tm) è la temperatura alla quale una metà del duplex di DNA si dissocia e diventa a singolo filamento. La stabilità del duplex DNA primer-template può essere misurata dalla temperatura di fusione (Tm).
4. Temperatura di ricottura ottimale (Ta Opt): La formula di Rychlik è la più rispettata. I nostri prodotti usano questa formula per calcolarla e migliaia di nostri clienti hanno riportato buoni risultati usandola per la fase di annealing del ciclo PCR. Di solito risulta in una buona resa del prodotto PCR con una produzione minima di prodotti falsi.
Ta Opt = 0,3 x(Tm del primer) + 0,7 x(Tm del prodotto) – 14.9
dove
Tm del primer è la temperatura di fusione della coppia primer-template meno stabile
Tm del prodotto è la temperatura di fusione del prodotto PCR.
5. Calcolo del mismatch Tm della coppia di primer: I due primer di una coppia di primer dovrebbero avere temperature di fusione molto simili per massimizzare la resa del prodotto PCR. La differenza di 5oC o più può portare a nessuna amplificazione.
Primer Design utilizzando il Software
Sono disponibili diversi strumenti di progettazione dei primer che possono assistere nella progettazione dei primer PCR sia per i nuovi utenti che per quelli esperti. Questi strumenti possono ridurre il costo e il tempo coinvolto nella sperimentazione, abbassando le possibilità di fallimento della sperimentazione.
Primer Premier segue tutte le linee guida specificate per la progettazione di primer PCR. Primer Premier può essere usato per progettare primer per modelli singoli, allineamenti, progettazione di primer degenerati, analisi degli enzimi di restrizione, analisi dei contig e progettazione di primer di sequenziamento.
Le linee guida per la progettazione di primer qPCR variano leggermente. Software come AlleleID e Beacon Designer possono progettare primer e sonde oligonucleotidiche per saggi di rilevazione complessi come i saggi multiplex, la progettazione di primer di specie incrociate, la progettazione di primer specifici di specie e la progettazione di primer per ridurre il costo della sperimentazione.
PrimerPlex è un software che può progettare primer per PCR multiplex e saggi multiplex di genotipizzazione SNP.