B. DNA-replikation i bakterier
I allmänhet replikeras DNA genom avveckling av spiralen, separation av strängen genom att vätebindningarna mellan de komplementära strängarna bryts och syntes av två nya strängar genom komplementär basparning. Replikationen börjar på en specifik plats i DNA:t som kallas replikationsursprunget (oriC).
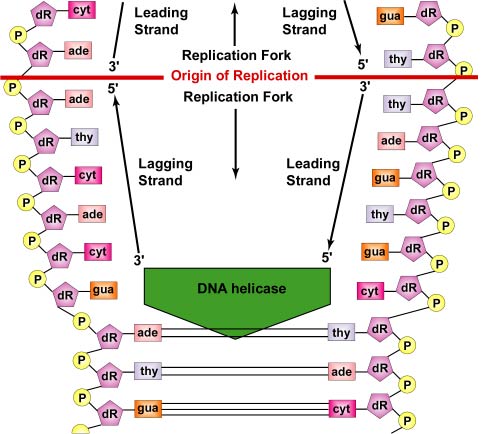
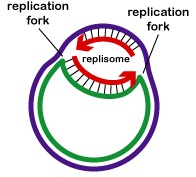
DNA-replikation är dubbelriktad från replikationsursprunget. För att påbörja DNA-replikationen får avvecklingsenzymer som kallas DNA-helikaser korta segment av de två föräldra-DNA-strängarna att avvecklas och separeras från varandra vid replikationens ursprung för att bilda två Y-formade replikationsgafflar. Dessa replikationsgafflar är själva platsen för DNA-kopiering (figur \(\PageIndex{3}\)). Alla proteiner som deltar i DNA-replikationen samlas vid replikationsgafflarna för att bilda ett replikationskomplex som kallas replisom (figur \(\PageIndex{4}\)).
Singelsträngsbindande proteiner binder sig till de enkelsträngade områdena så att de två strängarna inte kan återförenas. Avveckling av den dubbelsträngade helixen genererar positiva supercoils framför replikationsgaffeln. Enzymer som kallas topoisomeraser motverkar detta genom att producera brott i DNA:t och sedan återförena dem för att bilda negativa superspiraler för att lindra denna stress i den spiralformade molekylen under replikationen.
När strängarna fortsätter att spolas upp och separeras i båda riktningarna runt hela DNA-molekylen produceras nya komplementära strängar genom vätebindning av fria DNA-nukleotider med dem på varje föräldrasträng. När de nya nukleotiderna står på rad mittemot varje modersträng genom vätebindning förenar enzymer som kallas DNA-polymeraser nukleotiderna med hjälp av fosfodiesterbindningar. De nukleotider som står på rad genom komplementär basparning är faktiskt desoxynukleotidtrifosfater, som består av en kvävebaserad bas, desoxyribose, och tre fosfater. När fosfodiesterbindningen bildas mellan den nya nukleotikens 5′-fosfatgrupp och den sista nukleotikens 3′-OH i DNA-strängen avlägsnas två av fosfaterna, vilket ger energi för bindningen (se figur \(\PageIndex{6}\)). I slutändan tjänar varje modersträng som en mall för att syntetisera en komplementär kopia av sig själv, vilket resulterar i bildandet av två identiska DNA-molekyler (se figur \(\PageIndex{7}\)). I bakterier fungerar Par-proteiner för att separera bakteriekromosomer till motsatta poler i cellen under celldelningen. De binder sig till DNA:s replikationsursprung och drar eller trycker fysiskt isär kromosomerna, i likhet med mitotiska apparaten i eukaryota celler. Fts-proteiner, till exempel FtsK i divisomen, hjälper också till att separera den replikerade bakteriekromosomen.
GIF-animation som illustrerar DNA-replikation genom komplementär basparning
I verkligheten är DNA-replikation mer komplicerad än så på grund av DNA-polymerasernas natur. DNA-polymerasenzymerna kan bara koppla ihop fosfatgruppen vid 5′-kolet i en ny nukleotid med hydroxylgruppen (OH) vid 3′-kolet i en nukleotid som redan finns i kedjan. Därför kan DNA endast syntetiseras i en 5′- till 3′-riktning samtidigt som man kopierar en modersträng som löper i en 3′- till 5′-riktning.
Varje DNA-sträng har två ändar. DNA:s 5′-ände är den med den terminala fosfatgruppen på 5′-kolet i desoxyribosen; 3′-änden är den med en terminal hydroxylgrupp (OH) på desoxyribosen på 3′-kolet i desoxyribosen (se figur \(\PageIndex{8}\)). De två strängarna är antiparallella, det vill säga de löper i motsatt riktning. Därför kan den ena modersträngen – den som löper från 3′ till 5′ och kallas den ledande strängen – kopieras direkt i hela sin längd (se figur \(\PageIndex{9}\)). Den andra modersträngen – den som löper från 5′ till 3′ och kallas den efterföljande strängen – måste dock kopieras diskontinuerligt i korta fragment (Okazaki-fragment) på omkring 100-1000 nukleotider vardera när DNA:t rullas upp. Detta sker, som nämnts ovan, vid replisomen. Den eftersläpande DNA-strängen slingrar sig ut från den ledande strängen och detta gör det möjligt för replisomen att röra sig längs båda strängarna och dra igenom DNA:t när replikationen sker. Det är själva DNA:t, inte DNA-polymeraset, som rör sig under bakteriell DNA-replikation (se figur \(\PageIndex{5}\)).
DNA-polymerasenzymerna kan dessutom inte börja en ny DNA-kedja från grunden. De kan endast fästa nya nukleotider på 3′ OH-gruppen av en nukleotid i en redan existerande sträng. För att starta syntesen av den ledande strängen och varje DNA-fragment i den eftersläpande strängen krävs därför ett RNA-polymeraskomplex som kallas primas. Primaset, som kan sammanfoga RNA-nukleotider utan att kräva en befintlig nukleinsyrasträng, lägger först till flera kompletterande RNA-nukleotider mittemot DNA-nukleotiderna på huvudsträngen. Detta bildar en så kallad RNA-primer (se figur \(\PageIndex{10}\)).
DNA-polymeras III ersätter sedan primaset och kan lägga till DNA-nukleotider till RNA-primern (se figur \(\PageIndex{11}\)). Senare smälter DNA-polymeras II bort RNA-primern och ersätter RNA-nukleotiderna i primern med rätt DNA-nukleotider för att fylla luckan (se figur \(\PageIndex{12}\)). Slutligen kopplas DNA-fragmenten ihop av enzymet DNA-ligas (se figur \(\PageIndex{9}\)). Men trots detta komplicerade förfarande kan en 1 000 mikrometer lång makromolekyl av tätt packat, superlindat DNA göra en exakt kopia av sig själv på bara cirka 10 minuters tid under optimala förhållanden, genom att infoga nukleotider med en hastighet av cirka 1 000 nukleotider per sekund!
Animation av DNA-replikation.
Courtesy of HHMI’s Biointeractive.