Referenser >> PCR Primer
Riktlinjer för PCR Primer Design Guidelines
PCR (Polymerase Chain Reaction)
Polymerasekedjereaktionen anses allmänt vara en av 1900-talets viktigaste uppfinningar inom molekylärbiologin. Små mängder genetiskt material kan nu amplifieras för att kunna identifiera, manipulera DNA, upptäcka infektiösa organismer, inklusive de virus som orsakar aids, hepatit och tuberkulos, upptäcka genetiska variationer, inklusive mutationer, i mänskliga gener och många andra uppgifter.
PCR omfattar följande tre steg: Denaturering, glödgning och förlängning. Först denatureras det genetiska materialet, vilket omvandlar de dubbelsträngade DNA-molekylerna till enkelsträngar. Primerna glödgas sedan till de komplementära områdena i de enkelsträngade molekylerna. I det tredje steget förlängs de med hjälp av DNA-polymeraset. Alla dessa steg är temperaturkänsliga och de vanligaste temperaturerna är 94oC, 60oC respektive 70oC. En bra utformning av primerna är avgörande för lyckade reaktioner. De viktiga överväganden som beskrivs nedan är nyckeln till specifik amplifiering med hög avkastning. De rekommenderade värden som anges är inbyggda som standard i alla våra produkter.
1. Primerlängd: Det är allmänt accepterat att den optimala längden på PCR-primers är 18-22 bp. Denna längd är tillräckligt lång för adekvat specificitet och tillräckligt kort för att primrarna lätt ska kunna binda till mallen vid glödgningstemperaturen.
2. Primersmältningstemperatur: Primers smältningstemperatur (Tm) är per definition den temperatur vid vilken den ena halvan av DNA-duplexen dissocieras för att bli enkelsträngad och anger duplexens stabilitet. Primers med smältningstemperaturer i intervallet 52-58 oC ger i allmänhet de bästa resultaten. Primers med smältningstemperaturer över 65 oC har en tendens till sekundär glödgning. GC-innehållet i sekvensen ger en rättvis indikation på primerns Tm. Alla våra produkter beräknar den med hjälp av den termodynamiska teorin för närmaste granne, som är accepterad som en mycket bättre metod för att uppskatta den, och som anses vara den senaste och bästa tillgängliga.
Formel för beräkning av primerns Tm:
Smältningstemperatur Tm(K)={ΔH/ ΔS + R ln(C)}, eller smältningstemperatur Tm(oC) = {ΔH/ ΔS + R ln(C)}. – 273,15 där
ΔH (kcal/mol) : H är Enthalpin. Enthalpy är den mängd värmeenergi som ämnen har. ΔH är förändringen i Enthalpy. I ovanstående formel erhålls ΔH genom att addera alla di-nukleotidparens entalpivärden för varje närmast angränsande baspar.
ΔS (kcal/mol) : S är mängden oordning som ett system uppvisar kallas entropi. ΔS är förändringen av entropin. Här erhålls den genom att addera alla di-nukleotidpars entropivärden för varje närmast angränsande baspar. En ytterligare saltkorrigering läggs till eftersom parametrarna för närmaste grannar erhölls från DNA-smältningsstudier som utfördes i 1M Na+-buffert och detta är det standardvillkor som används för alla beräkningar.
ΔS (saltkorrigering) = ΔS (1M NaCl )+ 0.368 x N x ln()
Varvid
N är antalet nukleotidpar i primern ( primerlängd -1).
är saltekvivalent i mM.
beräkning:
= Koncentration av monovalenta joner +4 x fritt Mg2+.
3. Primer glödgningstemperatur: Primerns smälttemperatur är en uppskattning av DNA-DNA-hybridens stabilitet och kritisk för att bestämma glödgningstemperaturen. En för hög Ta ger otillräcklig hybridisering mellan primer och mall, vilket resulterar i ett lågt utbyte av PCR-produkter. För låg Ta kan möjligen leda till ospecifika produkter som orsakas av ett stort antal baspar-missmatchningar. Mismatch-tolerans har visat sig ha det starkaste inflytandet på PCR-specificiteten.
Ta = 0,3 x Tm(primer) + 0,7 Tm (produkt) – 14.9
varvid,
Tm(primer) = Smältningstemperatur för primrarna
Tm(produkt) = Smältningstemperatur för produkten
4. GC-innehåll: GC-innehållet (antalet G och C i primern i procent av de totala baserna) i primern bör vara 40-60 %.
5. GC-klämma: Närvaron av G- eller C-baser inom de sista fem baserna från 3′-ändan av primers (GC clamp) bidrar till att främja specifik bindning i 3′-ändan på grund av den starkare bindningen av G- och C-baser. Mer än tre G- eller C- baser bör undvikas i de sista fem baserna i primerns 3′-ändan.
6. Primerns sekundärstrukturer: Förekomst av sekundärstrukturer i primern som produceras av intermolekylära eller intramolekylära interaktioner kan leda till dåligt eller inget utbyte av produkten. De påverkar negativt annealing av primermallen och därmed amplifieringen. De minskar i hög grad tillgängligheten till primers i reaktionen.
i) Hårnålar: Den bildas genom intramolekylär interaktion inom primern och bör undvikas. Optimalt är en hårnål i 3′-ändan med ett ΔG på -2 kcal/mol och en inre hårnål med ett ΔG på -3 kcal/mol som i allmänhet tolereras.
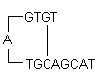
ΔG definition: Gibbs fria energi G är ett mått på hur mycket arbete som kan utvinnas ur en process vid konstant tryck. Den är ett mått på reaktionens spontanitet. Stabiliteten hos en hårnål representeras vanligen av dess ΔG-värde, den energi som krävs för att bryta sekundärstrukturen. Ett större negativt ΔG-värde indikerar stabila, oönskade hårnålar. Förekomst av hårnålar i 3′-ändan påverkar reaktionen mest negativt.
ΔG = ΔH – TΔS
ii) Självdimer: En primer-självdimer bildas genom intermolekylära interaktioner mellan två primers (samma mening), där primern är homolog till sig själv. I allmänhet används en stor mängd primers i PCR jämfört med mängden målgen. När primers bildar intermolekylära dimerer mycket lättare än att hybridisera med mål-DNA, minskar de produktavkastningen. Optimalt är att en 3′ end self dimer med ett ΔG på -5 kcal/mol och en intern self dimer med ett ΔG på -6 kcal/mol i allmänhet tolereras.
iii) Cross Dimer: Primer cross dimers bildas genom intermolekylär interaktion mellan sense- och antisense primers, där de är homologa. Optimalt är en 3′ end cross dimer med ett ΔG på -5 kcal/mol och en intern cross dimer med ett ΔG på -6 kcal/mol som i allmänhet tolereras.
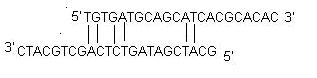
7. Upprepningar: En upprepning är en di-nukleotid som uppträder många gånger i följd och bör undvikas eftersom de kan misprime. Till exempel: EXEMPEL: ATATATATATAT. Det högsta tillåtna antalet upprepningar av di-nukleotider i en oligo är 4 di-nukleotider.
8. Löpningar: Primers med långa löpningar av en enda bas bör i allmänhet undvikas, eftersom de kan ge felprimering. Till exempel AGCGGGGGGGATGGGGGG har löpningar av basen ”G” med värdet 5 och 4. Maximalt antal körningar som accepteras är 4bp.
9. Stabilitet i 3′ ändar: Det är det maximala ΔG-värdet för de fem baserna från 3′-ändan. En instabil 3′-änd (mindre negativ ΔG) resulterar i mindre falsk priming.
10. Undvik mallens sekundära struktur: En enkelsträngad nukleinsyrasekvens är mycket instabil och viker sig i konformationer (sekundärstrukturer). Stabiliteten hos dessa sekundära strukturer beror till stor del på deras fria energi och smältningstemperaturer (Tm). Det är viktigt att ta hänsyn till sekundärstrukturer i mallen när man utformar primers, särskilt vid qPCR. Om primers utformas utifrån sekundärstrukturer som är stabila även över glödgningstemperaturerna, kan primers inte binda till mallen och utbytet av PCR-produkten påverkas avsevärt. Därför är det viktigt att utforma primers i de områden av mallen som inte bildar stabila sekundärstrukturer under PCR-reaktionen. Våra produkter fastställer sekundärstrukturerna i mallen och utformar primers som undviker dem.
11. Undvik korshomologi: För att förbättra primernas specificitet är det nödvändigt att undvika regioner med homologi. Primer som är utformade för en sekvens får inte amplifiera andra gener i blandningen. Vanligtvis utformas primers och sedan BLASTas för att testa specificiteten. Våra produkter erbjuder ett bättre alternativ. Du kan undvika regioner med korshomologi när du utformar primers. Du kan BLASTa mallarna mot en lämplig icke-redundant databas och programvaran tolkar resultaten. Den kommer att identifiera regioner med betydande korshomologier i varje mall och undvika dem under primersökningen.
Parametrar för utformning av primerpar
1. Amplikonlängd: Ampliklonlängden dikteras av de experimentella målen. För qPCR är mållängden närmare 100 bp och för standard-PCR är den nära 500 bp. Om man känner till positionerna för varje primer i förhållande till mallen beräknas produkten som: Produktlängd = (antisenseprimerns position – senseprimerns position) + 1.
2. Produktposition: Primer kan vara placerad nära 5′-ändan, 3′-ändan eller någonstans inom den angivna längden. Generellt sett är sekvensen nära 3′-ändan känd med större säkerhet och föredras därför oftast.
3. Produktens Tm: Smältningstemperatur (Tm) är den temperatur vid vilken den ena halvan av DNA-duplexen dissocieras och blir enkelsträngad. Stabiliteten hos primer-mall-DNA-duplexen kan mätas med hjälp av smältningstemperaturen (Tm).
4. Optimal glödgningstemperatur (Ta Opt): Rychliks formel är mest respekterad. Våra produkter använder denna formel för att beräkna den och tusentals av våra kunder har rapporterat goda resultat när de använder den för glödgningsteget i PCR-cykeln. Den resulterar vanligtvis i ett bra utbyte av PCR-produkter med minimal produktion av falska produkter.
Ta Opt = 0,3 x(Tm för primer) + 0,7 x(Tm för produkt) – 14.9
där
Tm för primer är smälttemperaturen för det mindre stabila primer-mallparet
Tm för produkt är smälttemperaturen för PCR-produkten.
5. Beräkning av primerparets Tm-mismatch: De två primrarna i ett primerpar bör ha nära överensstämmande smälttemperaturer för att maximera utbytet av PCR-produkter. En skillnad på 5oC eller mer kan leda till att ingen amplifiering sker.
Primerdesign med hjälp av programvara
Det finns ett antal verktyg för primerdesign som kan hjälpa både nya och erfarna användare att designa PCR-primer. Dessa verktyg kan minska kostnaden och tiden för experiment genom att minska risken för misslyckade experiment.
Primer Premier följer alla de riktlinjer som anges för PCR-primerdesign. Primer Premier kan användas för att designa primers för enskilda mallar, alignments, design av degenererade primers, restriktionsenzymanalys. contig-analys och design av primers för sekvensering.
Riktlinjerna för design av qPCR-primers varierar något. Programvara som AlleleID och Beacon Designer kan designa primers och oligonukleotidprober för komplexa detektionsanalyser, t.ex. multiplexanalyser, primerdesign för olika arter, primerdesign för specifika arter och primerdesign för att minska experimentkostnaderna.
rimerPlex är en programvara som kan designa primers för Multiplex PCR- och Multiplex SNP-genotypningsanalyser. rimerPlex är en programvara som kan designa primers för Multiplex PCR och Multiplex SNP genotyping assays.